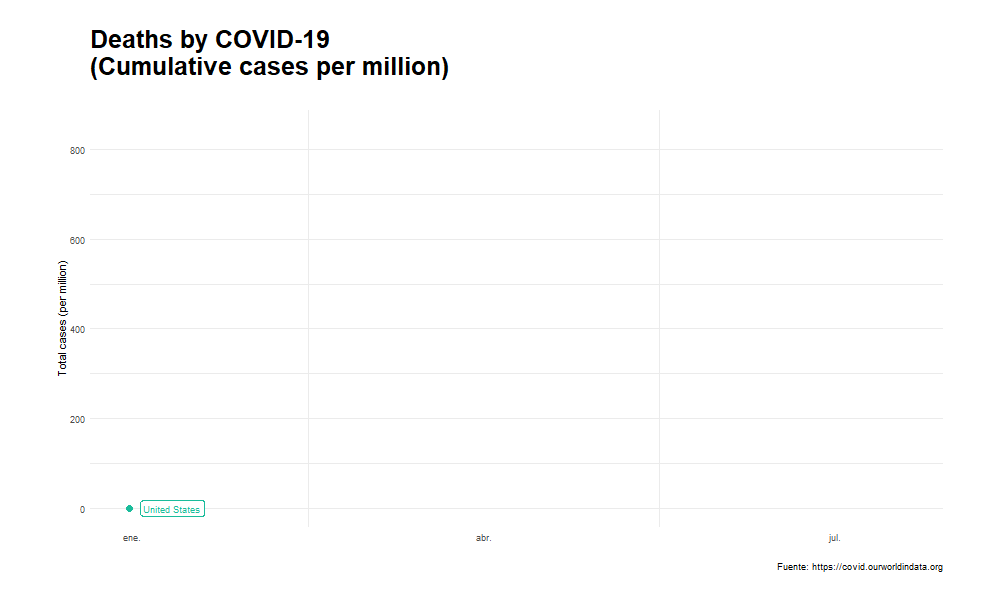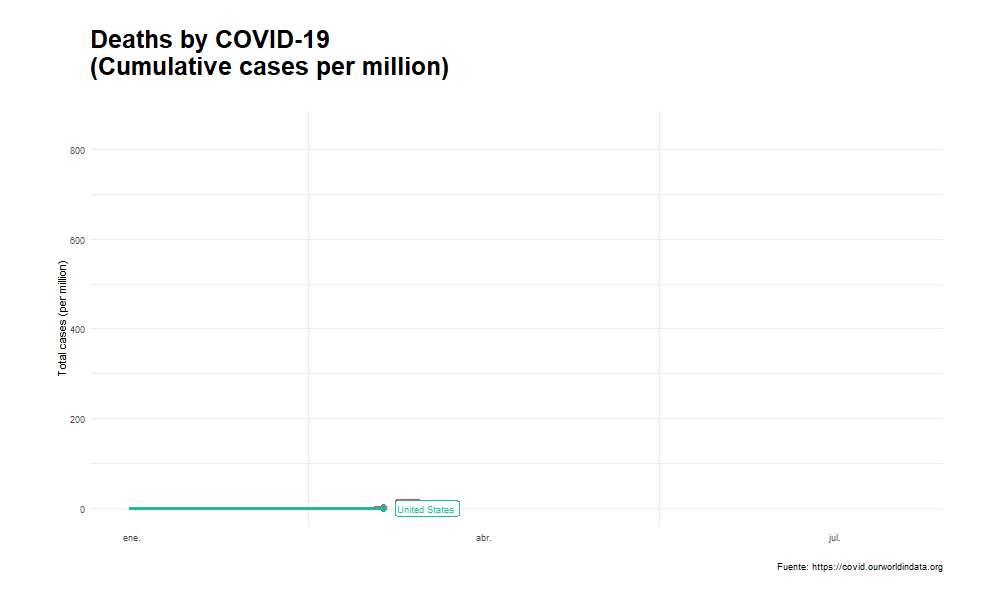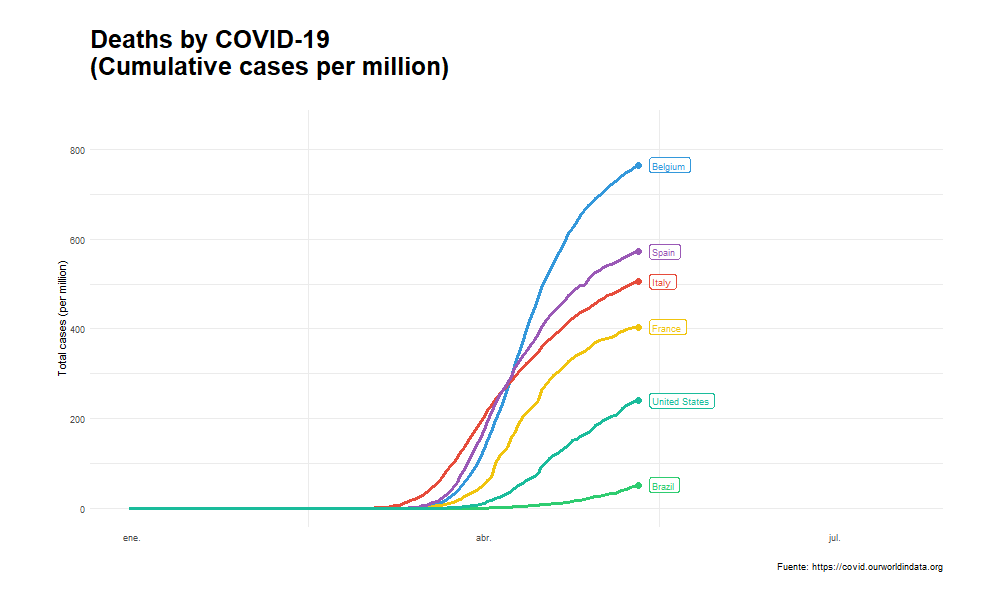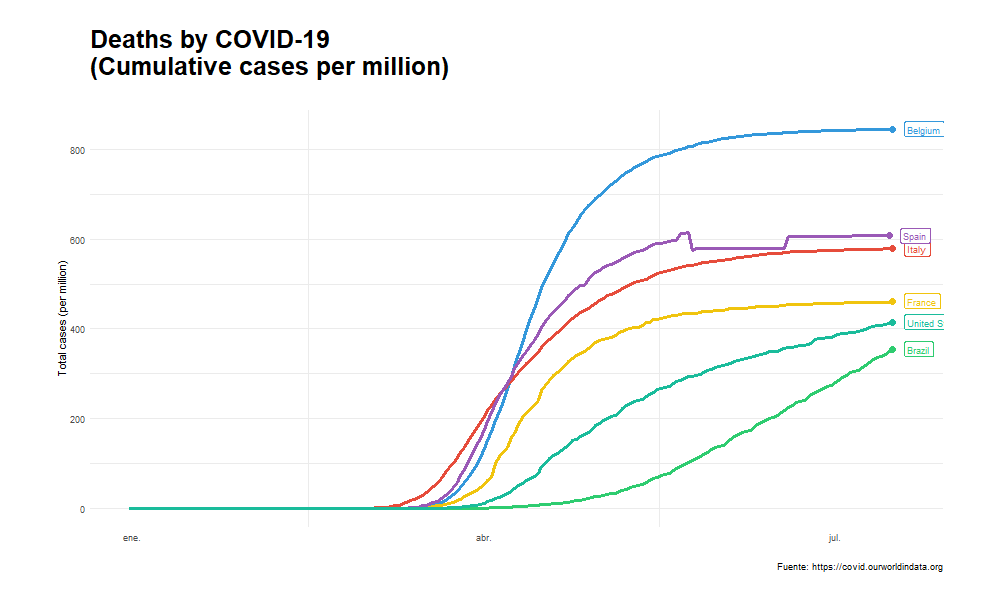Intro
Este post es, en cierta medida, una continuación del post anterior, donde presentamos varios gráficos animados utilizando {gganimate} y la función view_follow(). Para ejemplificar cómo realizar dichos gráficos utilizamos datos referentes al COVID-19 recopilados y actualizados diariamente por Our World in Data. En este segundo post se pretende explicar cómo añadir etiquetas (labels) a los gráficos animados con el paquete {gganimate} y, para ello, utilizaremos la misma fuente de datos.
En este post vamos a realizar dos tipos de gráficos animados. El primero de ellos será un gráfico de burbujas, una variable del gráfico de dispersión, donde cada burbuja representará un país de entre una lista de países seleccionados y donde el tamaño de las mismas será proporcional al tamaño en términos de población de dichos países. Cada burbuja irá acompañada de una etiqueta indicando qué país corresponde a cada una de las burbujas del gráfico. El segundo tipo de gráfico animado será un gráfico de líneas donde mostraremos la evolución de un grupo de países a lo largo del tiempo que lleva durando la pandemia. En este caso cada línea irá acompañada de una etiqueta que indicará el país corresponde a cada línea del gráfico. Vamos a ello!
Paquetes
library(tidyverse)
library(lubridate)
library(gganimate)
library(png)
library(gifski)
library(readxl)
library(ggthemr)
Dataset
df <- read_csv(url('https://raw.githubusercontent.com/owid/covid-19-data/master/public/data/owid-covid-data.csv'))
## Parsed with column specification:
## cols(
## .default = col_double(),
## iso_code = col_character(),
## continent = col_character(),
## location = col_character(),
## date = col_date(format = ""),
## total_tests = col_logical(),
## new_tests = col_logical(),
## total_tests_per_thousand = col_logical(),
## new_tests_per_thousand = col_logical(),
## new_tests_smoothed = col_logical(),
## new_tests_smoothed_per_thousand = col_logical(),
## tests_units = col_logical()
## )
## See spec(...) for full column specifications.
# theme:
ggthemr("flat")
opts <- theme_minimal() +
theme(panel.grid.major.x = element_blank(),
plot.title = element_text(size = 25,
face = "bold",
hjust = 0),
plot.subtitle = element_text(size=20,
face = "bold",
hjust = 0,
color = "orange"),
plot.margin= unit(c(t = 1, r = 1, b = 1, l = 2), "cm"))
1. Gráfico de Burbujas
Para este ejercicio hemos seleccionado un conjunto de países utilizando la función filter(), tal y como se puede observar en el código que sigue. Algunos de ellos los hemos seleccionado por curiosidad personal y otros de forma un poco aleatoria entre una selección provisional de países que registraban un número significativo de contagios. No existe por tanto una razón específica que justifique esta selección, que podría fácilmente haber sido cualquier otra y, por tanto, no esperamos inferir ninguna conclusión respecto a la evolución de los países seleccionados. Como hemos señalado, el objetivo del post es, básicamente, mostrar cómo agregar etiquetas a los gráficos. De hecho, en nuestro caso la lista de países que hemos seleccionado se encuentra en el código que mostramos abajo, pero si hubiéramos seguido algún tipo de patrón para la selección (por ejemplo filtrando los países con cierto número de casos, de fallecidos o de contagiados por millón de habitantes) no habría hecho falta escribir de forma manual los países elegidos y habría resultado incluso más sencillo de realizar.
df_paises <- df %>%
filter(
location == "United States" |
location == "Canada" |
location == "Mexico" |
location == "Brazil" |
location == "Peru" |
location == "Chile" |
location == "Argentina" |
location == "Ecuador" |
location == "Bolivia" |
location == "Colombia" |
location == "Dominican Republic" |
location == "Spain" |
location == "Portugal" |
location == "Germany" |
location == "Belgium" |
location == "Netherlands" |
location == "France" |
location == "Italy" |
location == "Sweden" |
location == "Turkey" |
location == "China" |
location == "South Korea" |
location == "Russia" |
location == "Ukraine" |
location == "Kazakhstan" |
location == "Pakistan" |
location == "India" |
location == "Bangladesh" |
location == "Saudi Arabia" |
location == "Qatar" |
location == "Oman" |
location == "Iran" |
location == "Iraq" |
location == "Egypt" |
location == "Morocco" |
location == "Algeria" |
location == "South Africa" |
location == "Nigeria" |
location == "Israel"
) %>%
group_by(date)
lbl_countries <- c("United States",
"Canada",
"Mexico",
"Brazil",
"Peru",
"Chile",
"Argentina",
"Ecuador",
"Bolivia",
"Colombia",
"Dominican Republic",
"Spain",
"Portugal",
"Germany",
"Belgium",
"Netherlands",
"France",
"Italy",
"Sweden",
"Turkey",
"China",
"South Korea",
"Brazil",
"Russia",
"Ukraine",
"Kazakhstan",
"Pakistan",
"India",
"Bangladesh",
"Saudi Arabia",
"Qatar",
"Oman",
"Iran",
"Iraq",
"Egypt",
"Morocco",
"Algeria",
"South Africa",
"Nigeria",
"Israel"
)
Una vez hemos seleccionado los países (df_paises) y hemos establecido qué etiquetas van a acompañar a cada uno de ellos (lbl_countries) procedemos a realizar el gráfico de burbujas. Utilizamos la función geom_text() para representar la fecha en el fondo del gráfico y geom_label() para las etiquetas de los países. El resultado será el siguiente:
x_paises <- df_paises %>%
ggplot(aes(x = total_deaths_per_million, y = total_cases_per_million, group = location)) +
geom_text(aes(label = as.factor(date)),
x= 400, y = 20000,
alpha = 0.5,
col = "gray80",
size = 40) +
geom_point(aes(size = population,
fill = continent),
alpha = 0.9,
shape = 21) +
geom_label(data = df_paises %>% filter(location %in% lbl_countries),
aes(total_deaths_per_million, total_cases_per_million, label = location),
nudge_x = -1) +
guides(size = "none") +
scale_size(range = c(10, 50)) +
transition_reveal(date) +
labs(title = "COVID-19 cases VS deaths by COVID-19 \n(Cumulative cases per million)",
subtitle= "",
y = "Total cases (per million)",
x = "Total deaths (per million)",
caption = "Fuente: https://covid.ourworldindata.org") +
opts
animate(x_paises, nframes= 400, width = 900, height= 900, fps = 20)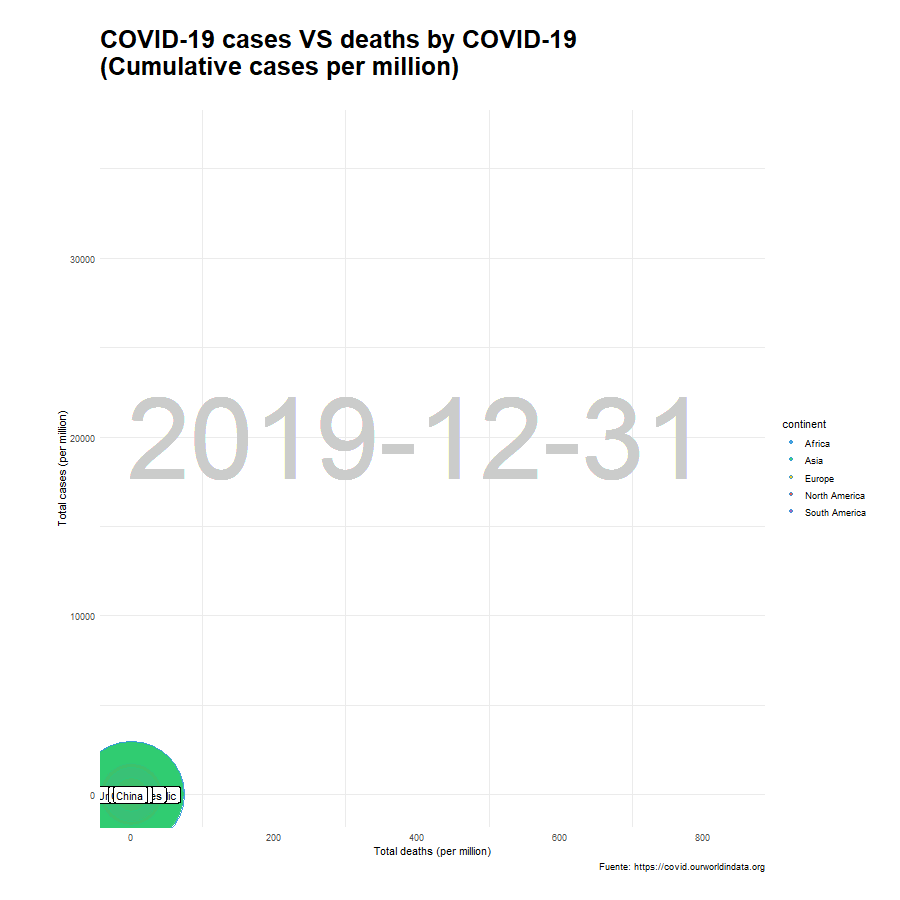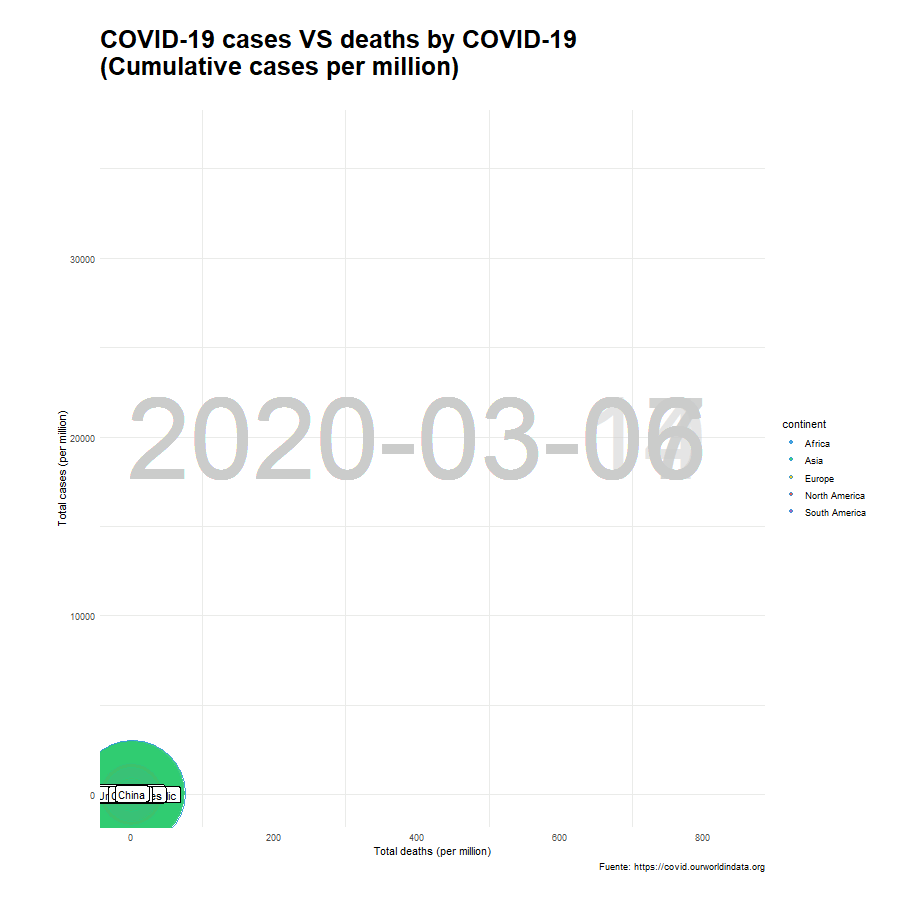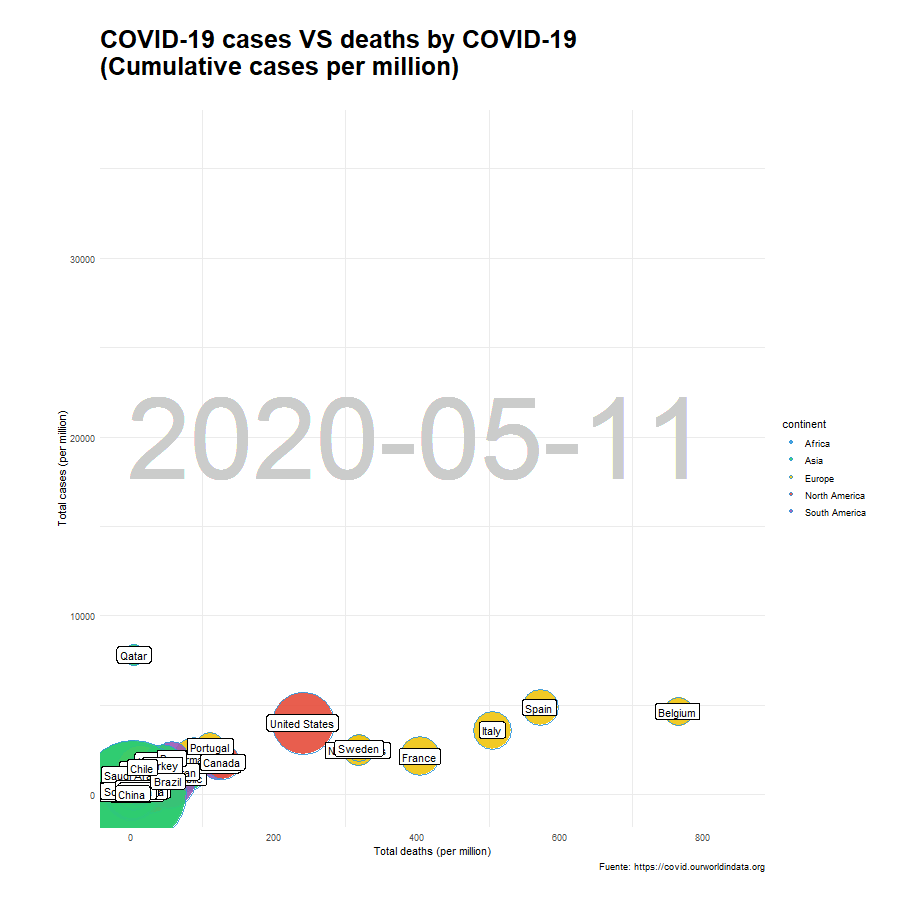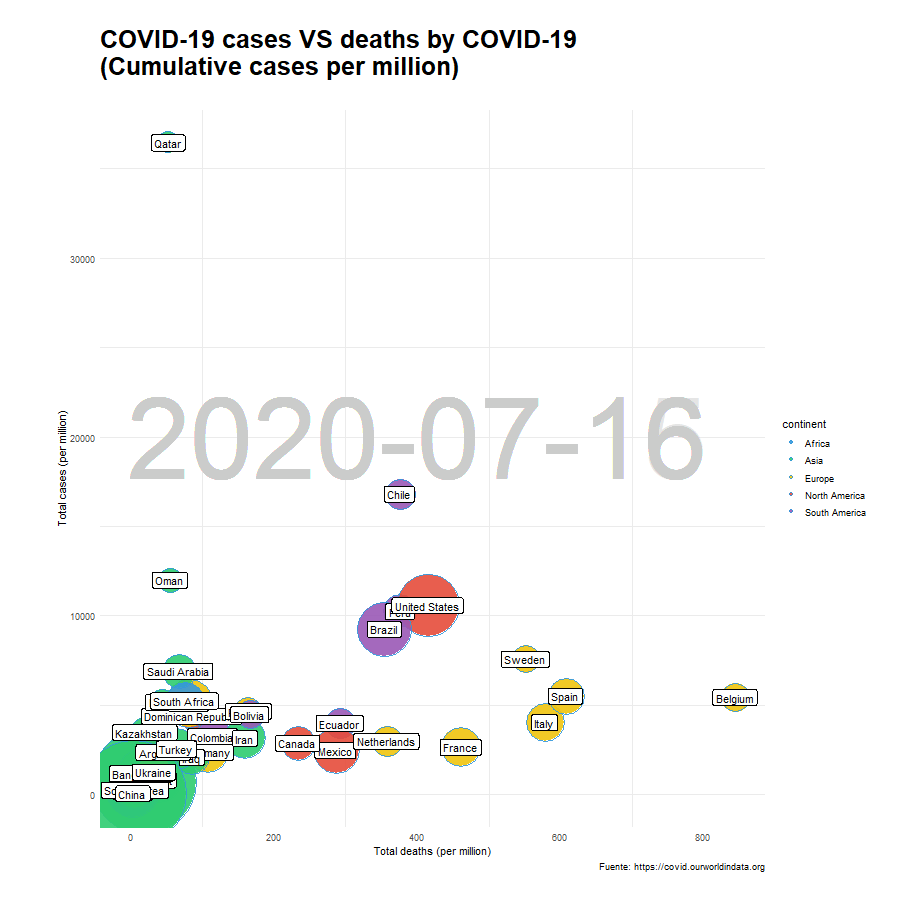
A modo de curiosidad añadimos un gráfico complementario utilizando una escala logarítmica para poder ver en mayor detalle lo que sucede al inicio del periodo, cuando los niveles de contagiios y fallecidos era relativamente pequeña. Nótese que los resultados comparan el número de contagiados y el número de fallecidos por millón de habitantes.
x_paises_log <- df_paises %>%
ggplot(aes(x = total_deaths_per_million, y = total_cases_per_million, group = location)) +
geom_text(aes(label = as.factor(date)),
x= 0.1, y = 0.9,
alpha = 0.5,
col = "gray80",
size = 40) +
geom_point(aes(size = population,
fill = continent),
alpha = 0.8,
shape = 21,
color = "grey80") +
geom_label(data = df_paises %>% filter(location %in% lbl_countries),
aes(total_deaths_per_million, total_cases_per_million, label = location)) +
scale_x_continuous(trans = 'log10', labels = scales::comma) +
scale_y_continuous(trans = 'log10', labels = scales::comma) +
guides(size = "none") +
scale_size(range = c(10, 50)) +
transition_reveal(date) +
labs(title = "COVID-19 cases VS deaths by COVID-19 \n(Cumulative cases per million)",
subtitle= "",
y = "Total cases (log scale)",
x = "Total deaths (log scale)",
caption = "Fuente: https://covid.ourworldindata.org") +
opts
animate(x_paises_log, nframes= 400, width = 900, height= 900, fps = 10)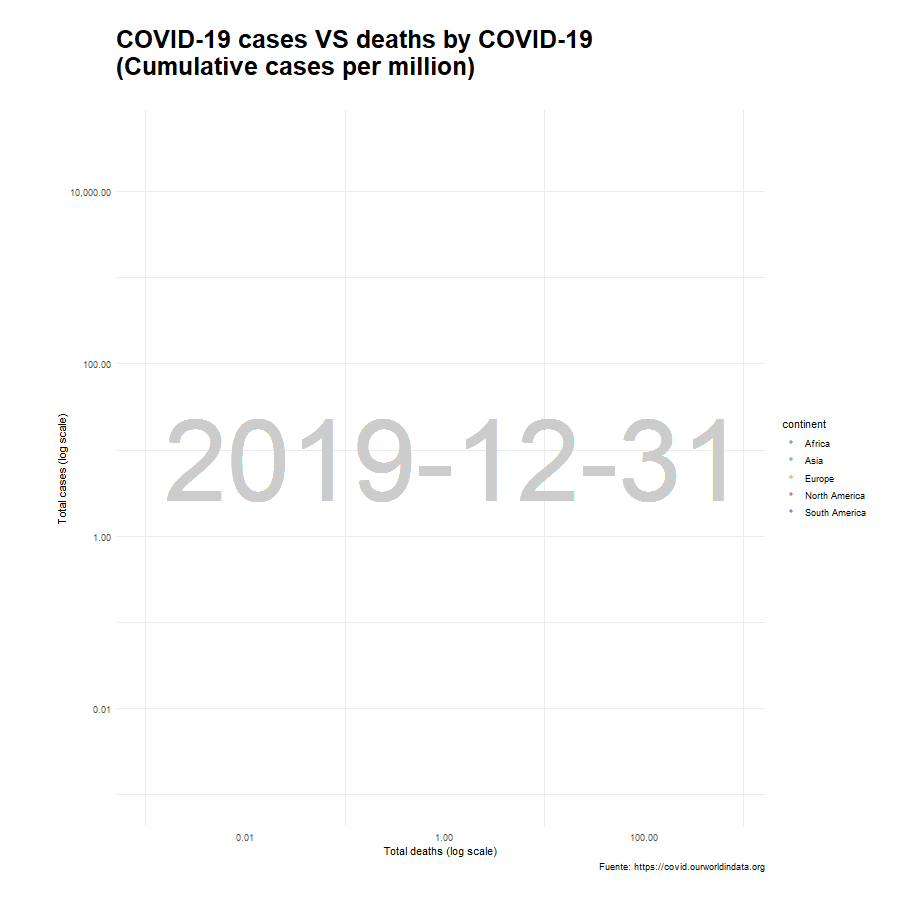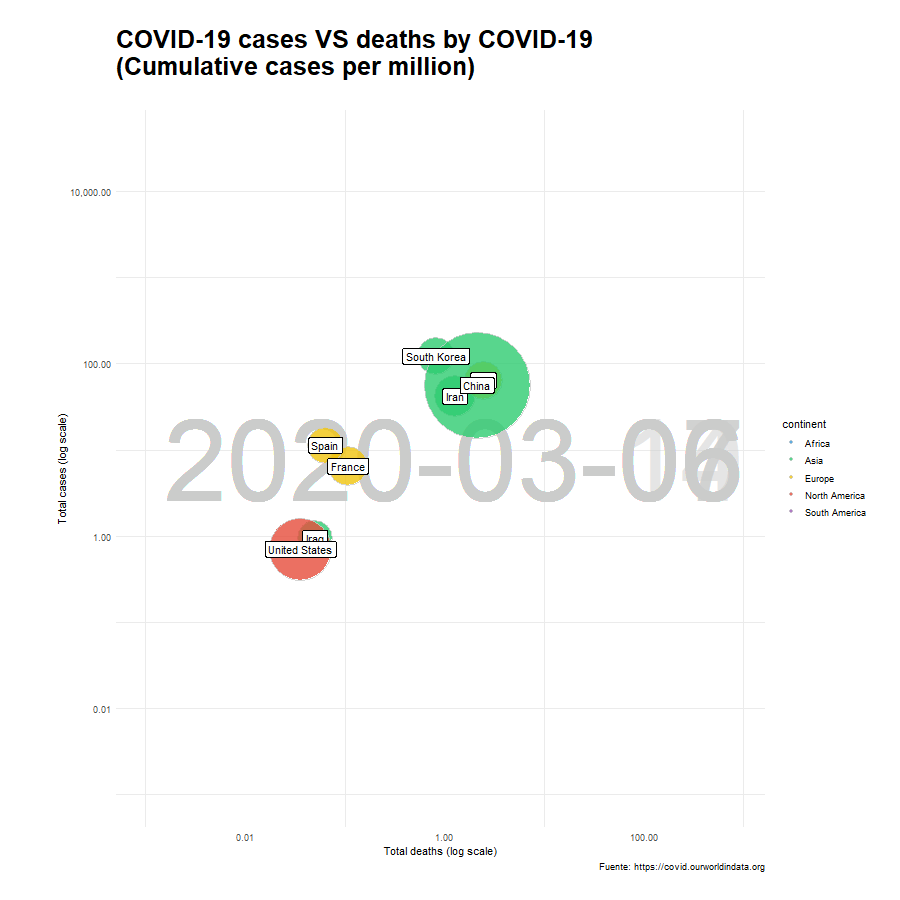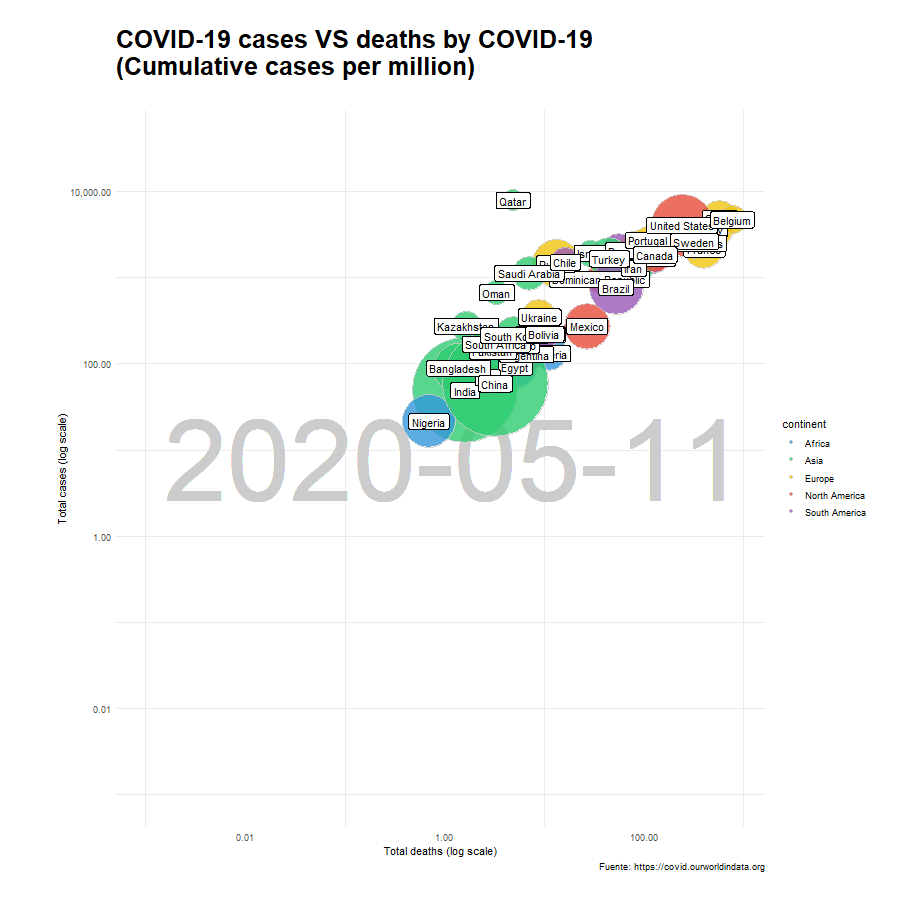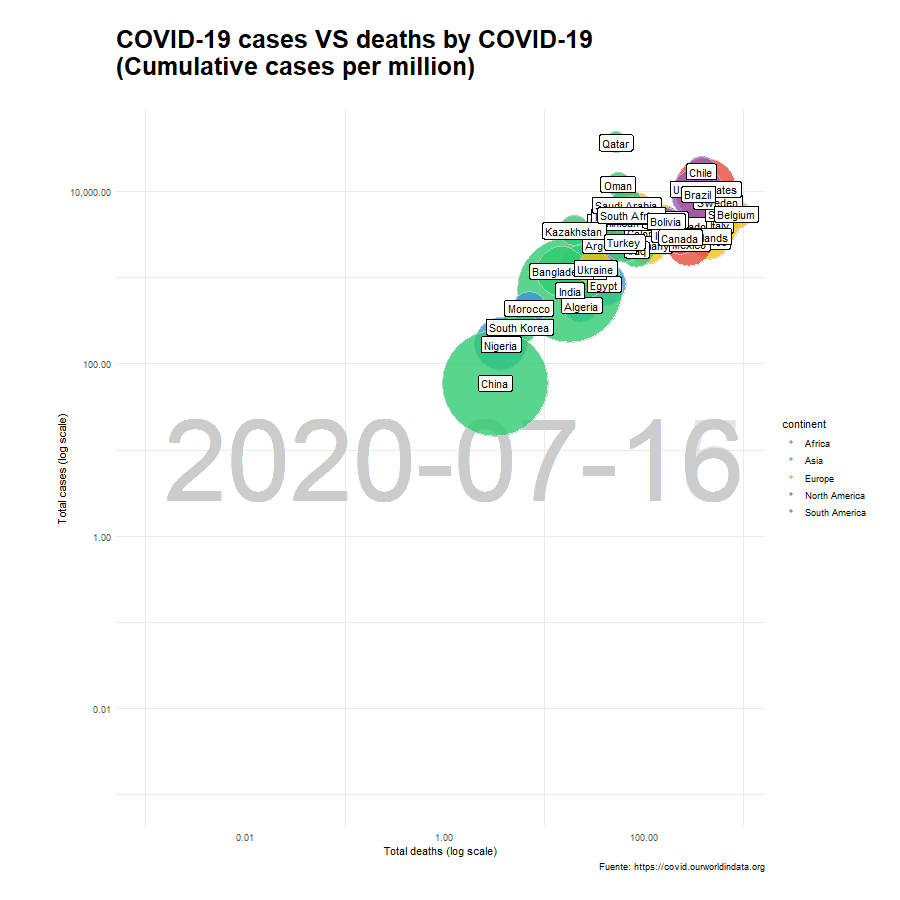
2. Gráfico de Línea
Generalmente utilizamos leyendas para indicar qué objetos del grafico (ya sean burbujas, líneas u otro) corresponde a cada variable que queremos representar. Una forma alternativa a las leyendas, especialmente en los gráficos de líneas, consiste en añadir etiquetas que acompañen a cada una de ellas. Esta alternativa funciona también en gráficos animados, aunque tenemos que asegurarnos que dichas etiquetas evolucionen conjuntamente con el resto del gráfico sin registrar cambios bruscos que hagan difícil su interpretación o empeoren la visualización. Para añadir estas etiquetas utilizaremos de nuevo la función geom_label() de forma análoga a cómo hemos hecho en los gráficos de burbujas realizados previamente.
df_lines <- df %>%
filter(location == "Spain" |
location == "Belgium" |
location == "Italy" |
location == "France" |
location == "United States" |
location == "Brazil") %>%
select(date, location, total_deaths_per_million)
x_lines <- df_lines %>%
ggplot(aes(x= date, y = total_deaths_per_million, color = location)) +
geom_line(size = 1.2) +
geom_point(size = 3) +
geom_label(aes(x= date, y = total_deaths_per_million, label = location),
nudge_x = 3,
size = 3,
hjust = 0) +
labs(title = "Deaths by COVID-19 \n(Cumulative cases per million)",
subtitle= "",
y = "Total cases (per million)",
x = "",
caption = "Fuente: https://covid.ourworldindata.org") +
transition_reveal(date) +
shadow_mark(past = FALSE) +
#view_follow(fixed_x = TRUE) +
enter_appear(early = FALSE) +
enter_grow() +
enter_fade(alpha = 0) +
theme_minimal() +
theme(panel.grid.major.x = element_blank(),
plot.title = element_text(size = 25,
face = "bold",
hjust = 0),
plot.subtitle = element_text(size=20,
face = "bold",
hjust = 0,
color = "orange"),
plot.margin= unit(c(t = 1, r = 2, b = 1, l = 2), "cm"),
legend.position = "none")
animate(x_lines, nframes= 400, width = 1000, height= 600, fps = 20)