Intro
El presente post tiene como objetivo presentar un conjunto de gráficos animados utilizando el paquete {gganimate} y la función view_follow(). El paquete {gganimate} está diseñado para realizar gráficos dinámicos (algunos ejemplos de gráficos animados con este paquete se pueden consultar aquí, aquí o aquí). Por su parte, la función view_follow() sirve para modificar los ejes x e y mientras avanza la animación, pudiendo con ello crear efectos dinámicos bastante interesantes.
Un aspecto que me llama la atención sobre la posibilidad de acercar los ejes de coordenadas al realizar un gráfico animado es que, con ello, el autor del gráfico podría en cierta medida influir en la percepción que se obtiene al observar el mismo. En gran medida somos conscientes que los datos pueden “estrujarse” hasta hacernos decir lo que queremos que digan o pueden interpretarse de distintas formas según el punto de vista desde el que sean analizados. Resulta evidente que unos podrían vender como un gran logro haber incrementado un 100% los ingresos anuales de su empresa, pasando de 10mil a 20mil euros en 12 meses, mientras que ante los mismos datos otros podrían criticar el resultado empresarial argumentando que el promedio de ingresos anual en la década pasada rondaba los 50mil o que su principal competidor factura 20mil en un solo mes sin gran esfuerzo. En cierta medida algo parecido podría tener lugar a la hora de representar información en un gráfico, principalmente cuando el gráfico es animado y, especialmente, cuando la perspectiva del mismo en función de los ejes puede ser manipulada.
En este ejercicio los ejemplos los realizaremos utilizando datos referentes al COVID-19 actualizados por Our World in Data. Hasta el momento este dataset se actualiza diariamente e incluye información sobre casos confirmados y fallecimientos por COVID19, sobre pruebas de diagnóstico y otras variables de interés.
Paquetes
library(tidyverse)
library(lubridate)
library(gganimate)
library(png)
library(gifski)
library(readxl)
library(viridis)
library(ggthemr)
library(ggrepel)
Dataset
df <- read_csv(url('https://raw.githubusercontent.com/owid/covid-19-data/master/public/data/owid-covid-data.csv'))
## Parsed with column specification:
## cols(
## .default = col_double(),
## iso_code = col_character(),
## continent = col_character(),
## location = col_character(),
## date = col_date(format = ""),
## total_tests = col_logical(),
## new_tests = col_logical(),
## total_tests_per_thousand = col_logical(),
## new_tests_per_thousand = col_logical(),
## new_tests_smoothed = col_logical(),
## new_tests_smoothed_per_thousand = col_logical(),
## tests_units = col_logical()
## )
## See spec(...) for full column specifications.
## Warning: 69184 parsing failures.
## row col expected actual file
## 1026 total_tests 1/0/T/F/TRUE/FALSE 13330.0 <connection>
## 1026 total_tests_per_thousand 1/0/T/F/TRUE/FALSE 0.295 <connection>
## 1026 tests_units 1/0/T/F/TRUE/FALSE tests performed <connection>
## 1027 total_tests 1/0/T/F/TRUE/FALSE 14850.0 <connection>
## 1027 new_tests 1/0/T/F/TRUE/FALSE 1520.0 <connection>
## .... ........................ .................. ............... ............
## See problems(...) for more details.
En primer lugar vamos a trabajar con datos globales (a nivel mundial) y, por tanto, para obtener solo datos agregados filtraremos el dataset y seleccionaremos solo la información referente a World. Por su parte vamos a seleccionar las variables new_cases, total_cases, total_deaths y new_deaths, que serán las variables a representar, y a continuación agruparemos la información por fecha. Nuestro nuevo dataset será, por tanto, el siguiente:
df_world <- df %>%
filter(location == "World") %>%
select(date, location, new_cases, total_cases, new_deaths, total_deaths) %>%
group_by(date)
1. Evolución del número diario de casos confirmados por COVID-19
1.1. Gráfico básico sin animación
Empezamos realizando un gráfico estático construido utilizando {ggplot2} donde se muestre la evolución del número total de casos confirmados por COVID-19 hasta la fecha. Para mayor información sobre cómo realizar gráficos de barras con {ggpplot2} les invito a visitar este post.
opts <- theme_minimal() +
theme(panel.grid.major.x = element_blank(),
plot.title = element_text(size = 25,
face = "bold",
hjust = 0),
plot.subtitle = element_text(size=20,
face = "bold",
hjust = 0,
color = "orange"),
plot.margin= unit(c(t = 1, r = 1, b = 1, l = 2), "cm"))
update <- format(max(df_world$date), "%Y-%m-%d")
df_world %>%
ggplot(aes(x = date,
y = new_cases,
fill = "orange")) +
geom_col() +
guides(fill = FALSE) +
scale_y_continuous(labels = scales::comma) +
labs( x= "",
y= " ",
title = "Confirmed COVID-19 cases \n(Daily change)",
subtitle = bquote('('~Updated==.(update)~')'),
caption = "Source: https://covid.ourworldindata.org") +
opts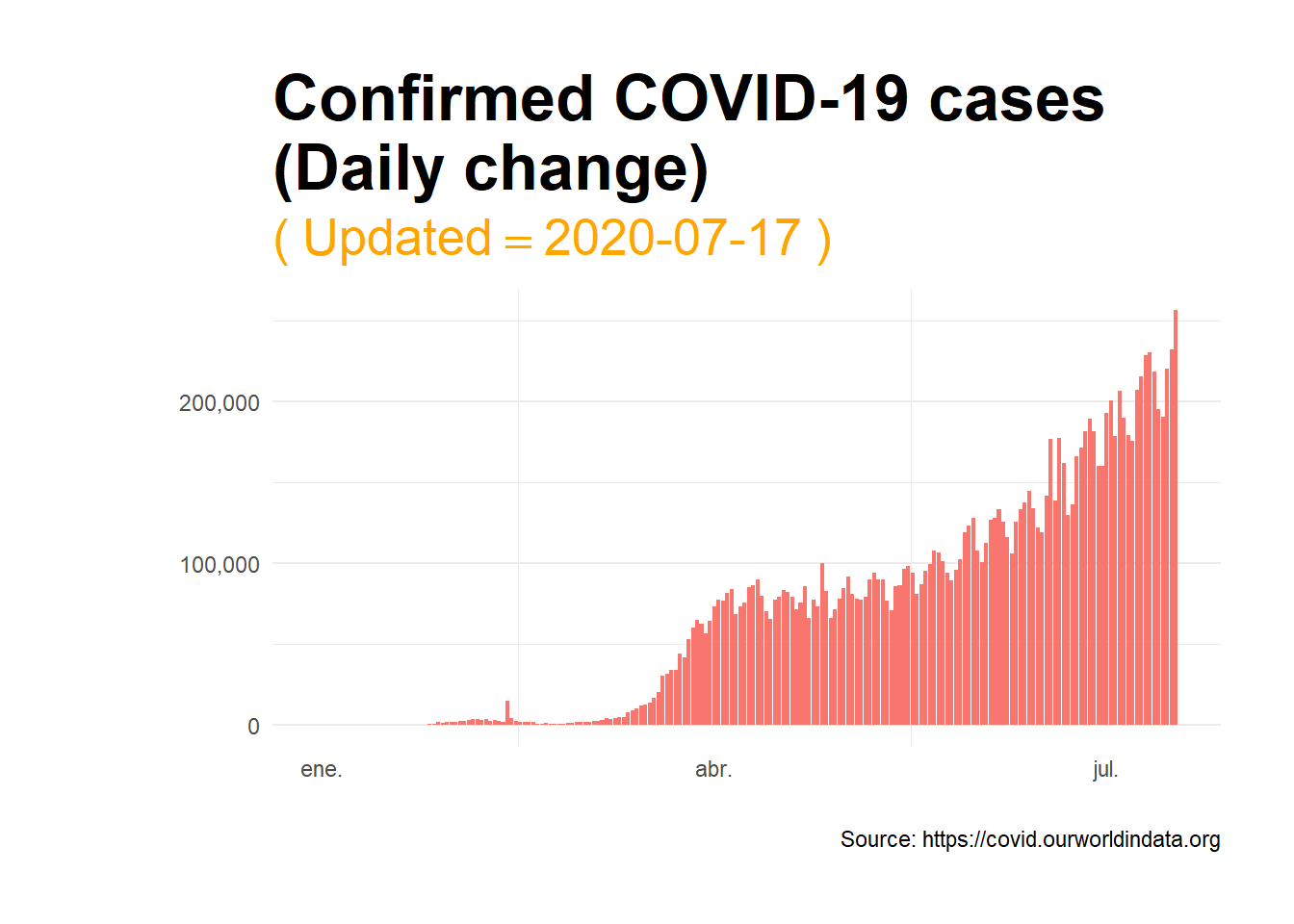
El número de casos confirmados diarios mantiene una clara tendencia ascendente, reforzando la opinión de la Organización Mundial de la Salud (WHO) cuando asegura que estamos lejos de haber terminado con la pandemia (“Coronavirus pandemic is far from over”).
1.2. Gráfico animado con follow_view()
Si dinamizamos el gráfico anterior y aplicamos la función follow_view() obtenemos un gráfico animado donde los ejes x e y van variando progresivamente según va evolucionando el periodo de tiempo que estamos examinando. Sin embargo, como señalábamos previamente, un gráfico de estas características puede ofrecer cierta sensación de gran intensidad en el crecimiento del número de casos confirmados en ciertos periodos, perdiéndose un poco la perspectiva general de la evolución del indicador. Una animación de este estilo puede ser especialmente un tanto inapropiada cuando se registran fuertes cambios entre un periodo y otro, como sucede a mediados del mes de febrero, donde encontramos cambios muy bruscos en la animación.
plot_animated <- df_world %>%
ggplot(aes(x = date,
y = new_cases,
fill = "orange")) +
geom_col() +
guides(fill = FALSE) +
scale_y_continuous(labels = scales::comma) +
labs( x= "",
y= " ",
title = "Confirmed COVID-19 cases \n(Daily change)",
subtitle = "Date : {closest_state}",
caption = "Source: https://covid.ourworldindata.org") +
transition_states(date, wrap = F) +
shadow_mark() +
enter_appear() +
enter_grow() +
enter_fade() +
view_follow() +
opts
animate(plot_animated, nframes= 400, width = 1000, height= 600, fps = 7)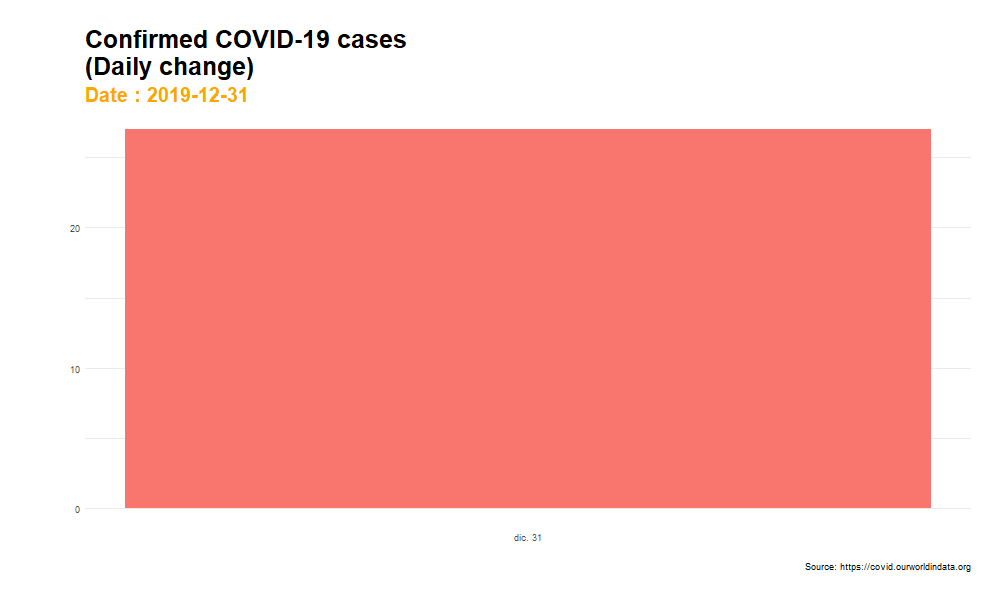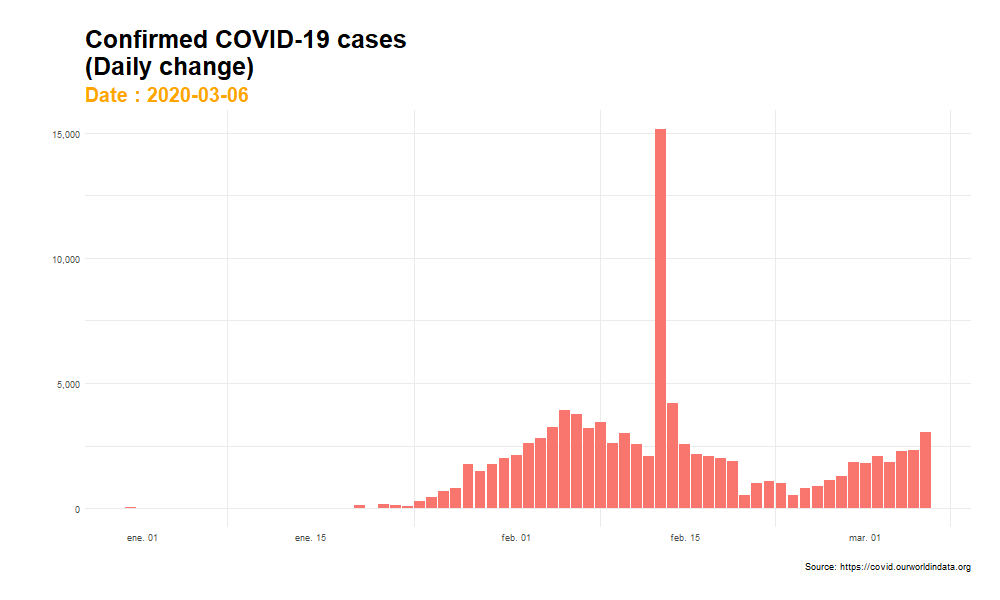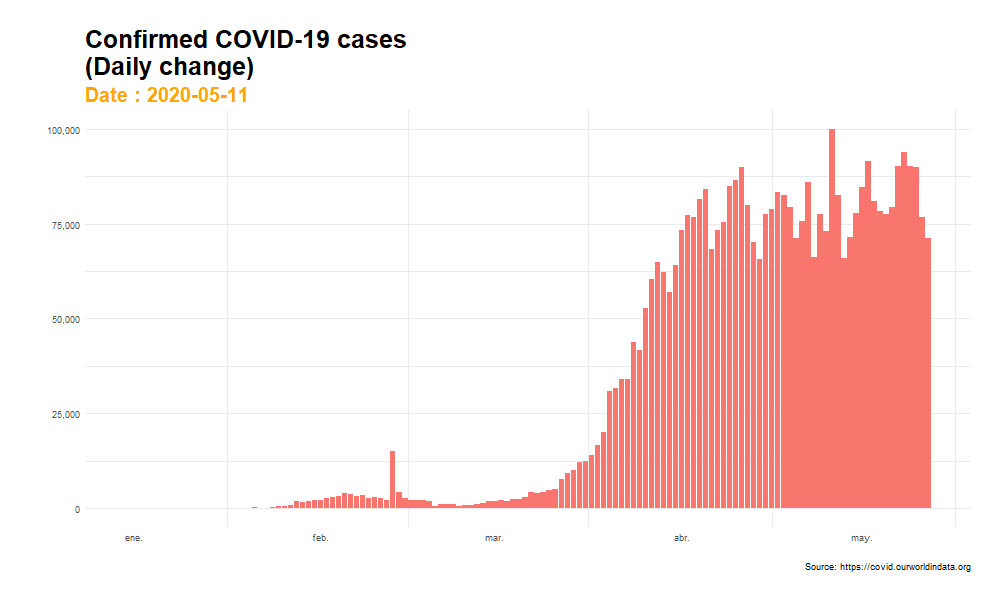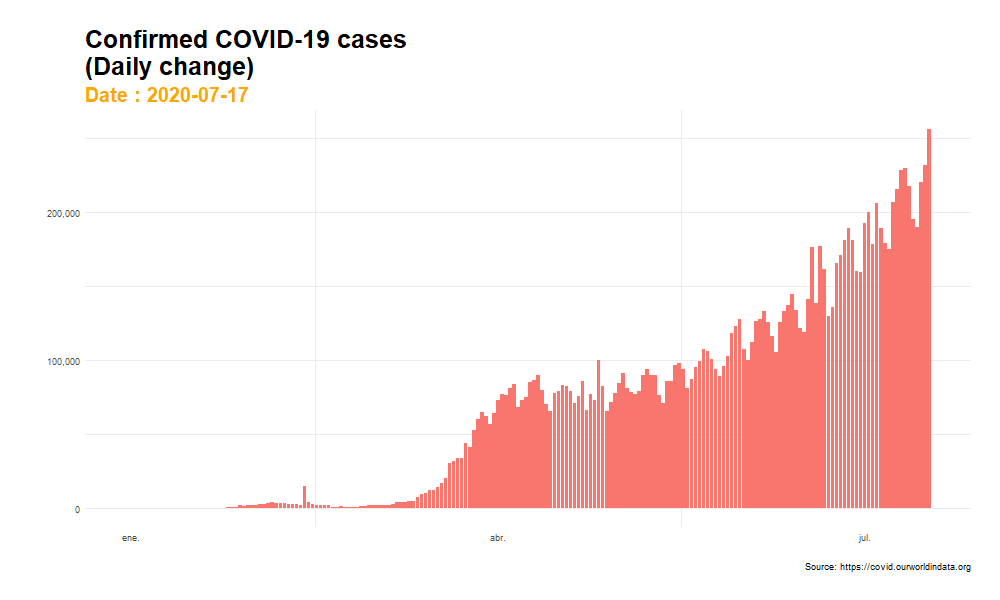
2. Evolución del número diario de fallecidos por COVID-19.
2.1. Gráfico básico sin animación
La evolución del triste número diario de personas fallecidas debido al COVID-19 se muestra en el siguiente gráfico. A diferencia de lo que sucede con el número diario de contagios, cantidad que sigue incrementándose con el paso del tiempo, el número de fallecidos ha registrado cierta tendencia descendente desde los meses de abril y mayo, donde se alcanzó un evidente pico. No obstante, también se observa un claro estancamiento de dicha tendencia descendente en el número de muertos y cierta incertidumbre con respecto al futuro de la pandemia en estos términos. Además, el número de fallecidos diarios continúa siendo elevado y, por tanto, a pesar de los procesos de deconfinamiento que se están dando en muchos países no es de extrañar que el coronavirus deba continuar siendo un motivo de gran preocupación para todos.
df_world %>%
ggplot(aes(x = date,
y = new_deaths,
fill = "orange")) +
geom_col() +
guides(fill = FALSE) +
scale_y_continuous(labels = scales::comma) +
labs( x= "",
y= " ",
title = "Deaths by COVID-19 \n(Daily change)",
subtitle = bquote('('~Updated==.(update)~')'),
caption = "Source: https://covid.ourworldindata.org") +
opts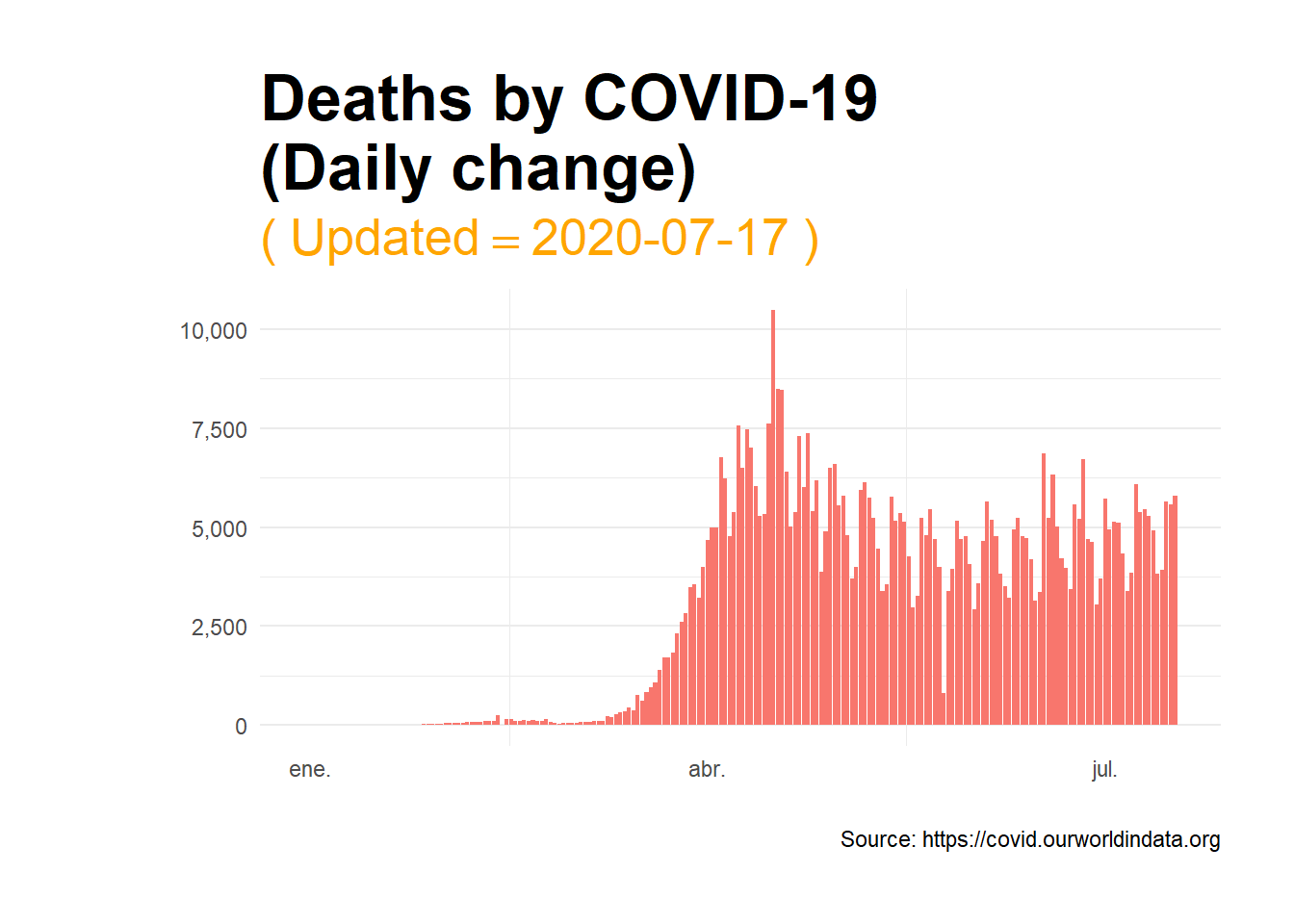
2.2. Gráfico animado con eje x fijo
Cómo dijimos previamente, en aquellos casos donde nos encontremos con cambios bruscos en la evolución de los datos puede que un gráfico animado como el anterior no resulte del todo adecuado. Puede que en casos de dichas características convenga fijar el eje x, el eje y o incluso ambos. Veamos cómo quedaría el grafico anterior, en el que se muestra la evolución del número diario de fallecidos, fijando el eje x.
plot_animated <- df_world %>%
ggplot(aes(x = date,
y = new_deaths,
fill = "orange")) +
geom_col() +
guides(fill = FALSE) +
scale_y_continuous(labels = scales::comma) +
labs( x= "",
y= " ",
title = "Deaths by COVID-19 \n(Daily change)",
subtitle = "Date : {closest_state}",
caption = "Source: https://covid.ourworldindata.org") +
transition_states(date, wrap = F) +
shadow_mark() +
enter_appear() +
enter_grow() +
enter_fade() +
view_follow(fixed_x = TRUE) +
opts
animate(plot_animated, nframes= 400, width = 1000, height= 600, fps = 7)
3. Evolución del número acumulado de casos confirmados por COVID-19
3.1. Gráfico básico sin animación
El número acumulado de casos de COVID-19 confirmados registró un importante cambio de tendencia desde los meses de marzo y abril, periodo en el esta cantidad se empezó a acelerarse notablemente. El gráfico de línea estático que se muestra a continuación refleja claramente esta evolución. Además, dicho gráfico sugiere que el número de casos está incrementándose más rápidamente, lo que posiblemente tenga su explicación en la expansión de la enfermedad en numerosos países y debido a la realización de más pruebas para la detección del mismo.
df_world %>%
ggplot(aes(x = date,
y = total_cases,
fill = "orange")) +
geom_line(color = "orange",
size = 1) +
geom_point(color = "orange",
size = 1.5) +
guides(fill = FALSE) +
scale_y_continuous(labels = scales::comma) +
labs( x= "",
y= " ",
title = "Confirmed COVID-19 cases \n(Cumulative cases)",
subtitle = bquote('('~Updated==.(update)~')'),
caption = "Source: https://covid.ourworldindata.org") +
opts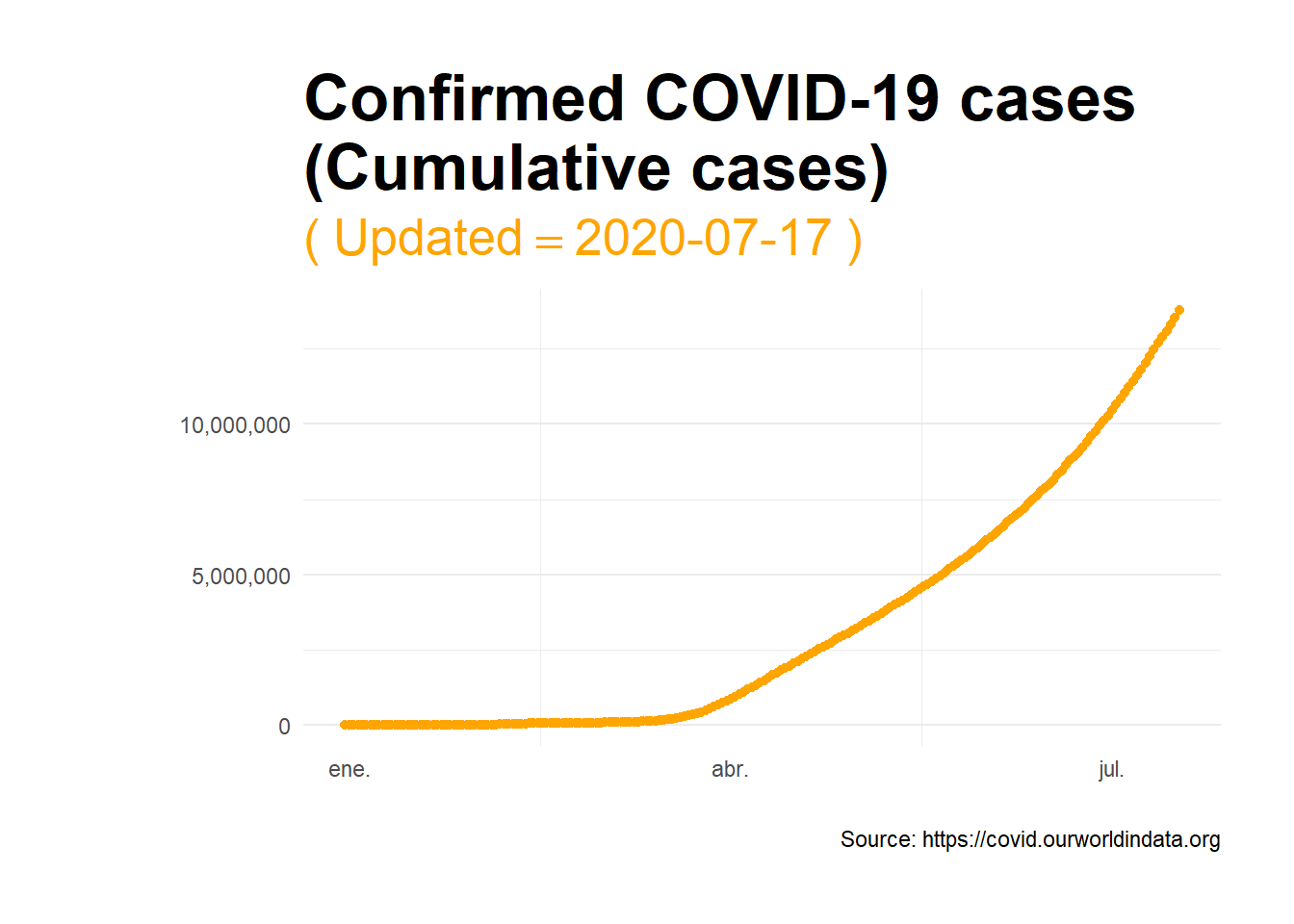
3.2. Gráfico animado con follow_view()
El gráfico anterior nos permite observar la evolución de los casos acumulados por COVID-19 en su conjunto, es decir, la visión global de la evolución desde el inicio de la epidemia hasta la fecha actual. Sin embargo, dado que esta cifra ha incrementado de forma abismal durante los meses pasados, especialmente desde los meses de marzo y abril, dicho gráfico refleja la tendencia general, y la cantidad alcanzada, pero es incapaz de mostrarnos en mayor detalle los cambios de menor magnitud que han ido sucediéndose a lo largo de cada mes. Para poder reflejar dichas variaciones de menor tamaño puede resultar de gran utilidad la animación del gráfico y el uso de la función view_follow(). No obstante, la animación resultante, que en cierta medida recuerda a una serpiente avanzando rápidamente en busca de su presa, puede añadir cierta sensación de dramatismo al dramatismo que ya de por sí supone el rápido incremento que ha tenido, y está teniendo, el número de personas contagiadas durante esta pandemia.
plot_animated_2 <- df_world %>%
ggplot(aes(x = date,
y = total_cases)) +
geom_bar(stat= "identity",
color = "white",
fill = "grey90",
show.legend = FALSE) +
geom_line(color = "orange",
size = 1.5) +
geom_point(color = "orange",
size = 4) +
geom_text(aes(x= min(date), y = min(total_cases), label = as.factor(date)),
hjust = -0.05,
vjust = -3,
alpha = 0.5,
col = "gray80",
size = 40) +
transition_reveal(date) +
view_follow() +
enter_appear() +
shadow_mark() +
enter_grow() +
enter_fade() +
guides(fill = FALSE) +
scale_y_continuous(labels = scales::comma) +
labs(title = "Confirmed COVID-19 cases \n(Cumulative cases)",
subtitle = "",
y = "",
x = "",
caption = "Fuente: https://covid.ourworldindata.org") +
opts
animate(plot_animated_2, nframes= 400, width = 1000, height= 600, fps = 7)
4. Evolución del número acumulado de fallecidos por COVID-19
4.1. Gráfico básico sin animación
La evolución que sin duda produce una mayor tristeza es la que hace referencia al número acumulado total de personas fallecidas a causa de la pandemia hasta el momento. Esta evolución se muestra en el gráfico estático que sigue a continuación. La línea continúa mostrando un progresivo incremento y no parece que en las últimas semanas haya tenido lugar una significativa reducción de la tendencia registrada desde los meses de abril y mayo.
df_world %>%
ggplot(aes(x = date,
y = total_deaths,
fill = "orange")) +
geom_line(color = "orange",
size = 1) +
geom_point(color = "orange",
size = 1.5) +
guides(fill = FALSE) +
scale_y_continuous(labels = scales::comma) +
labs( x= "",
y= " ",
title = "Deaths by COVID-19 \n(Cumulative cases)",
subtitle = bquote('('~Updated==.(update)~')'),
caption = "Source: https://covid.ourworldindata.org") +
opts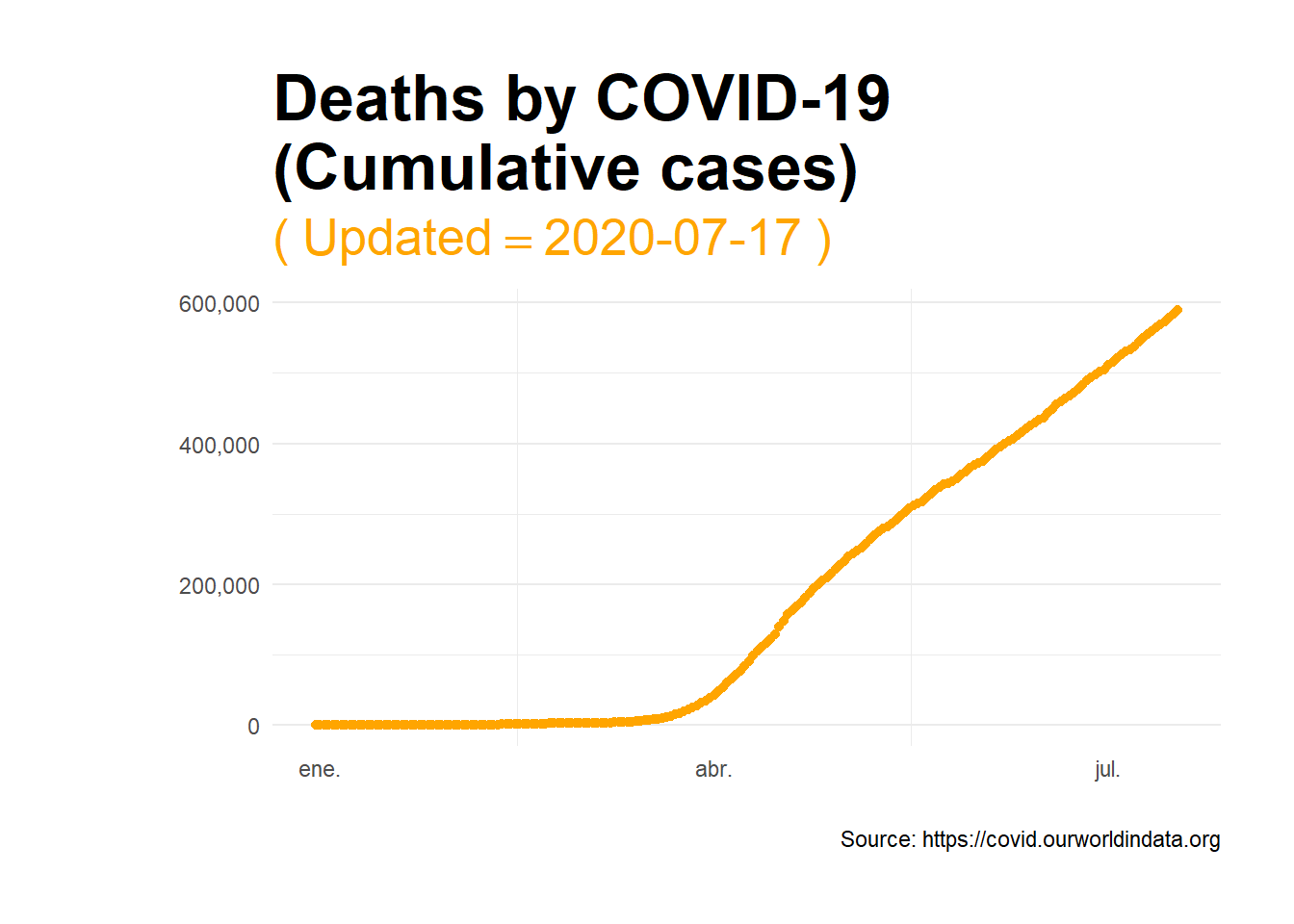
4.2. Gráfico animado con con eje x fijo
Veamos, por último, cómo sería el resultado que obtendríamos al representar de forma animada el gráfico anterior fijando de nuevo el eje x. En este caso, debido a que al ser cifras acumuladas el valor del eje y no deja de incrementarse, el resultado obtenido se asemeja al de una cobra real preparada para atacar. En aquellos momentos donde el incremento ha sido mayor la línea muestra una mayor verticalidad, mientras que, como cabe esperar, una mayor horizontalidad de la misma será indicativo de un decrecimiento en el número de fallecidos por el virus.
plot_animated_2 <- df_world %>%
ggplot(aes(x = date,
y = total_deaths)) +
geom_bar(stat= "identity",
color = "white",
fill = "grey90",
show.legend = FALSE) +
geom_line(color = "orange",
size = 1.5) +
geom_point(color = "orange",
size = 4) +
geom_text(aes(x= min(date), y = min(total_deaths), label = as.factor(date)),
hjust = -0.05,
vjust = -3,
alpha = 0.5,
col = "gray80",
size = 40) +
transition_reveal(date) +
view_follow(fixed_x = TRUE) +
enter_appear() +
shadow_mark() +
enter_grow() +
enter_fade() +
guides(fill = FALSE) +
scale_y_continuous(labels = scales::comma) +
labs(title = "Deaths by COVID-19 \n(Cumulative cases)",
subtitle = "",
y = "",
x = "",
caption = "Fuente: https://covid.ourworldindata.org") +
opts
animate(plot_animated_2, nframes= 400, width = 1000, height= 600, fps = 7)