Paquetes
En primer lugar descargamos el paquete {sqldf} y el paquete {tidyverse} que nos servirá para comparar la sintaxis de SQL con la sintaxis tradicional de {dplyr}. Téngase en cuenta que en este post anterior se expone en mayor profundidad las principales funcionalidades del paquete {tidyverse}.
library(sqldf)
library(tidyverse)
library(gapminder)
library(Hmisc)
Introducción
En este segundo post, que continúa la exposición que iniciamos en el post previo de cómo trabajar con la sintaxis de SQL en R utilizando el paquete {sqldf}, vamos a explicar cómo unir tablas y cómo compilar información de diferentes fuentes.
Compilar información proveniente de varias tablas es un aspecto fundamental en el análisis de datos. Aunque a día de hoy todavía no hemos realizado un post específico explicando cómo hacer estas operaciones en R, en varios post previos hemos tenido que realizar estas operaciones (véase por ejemplo aquí, aquí o aquí). Por todo ello, aunque el objetivo principal de este post consiste en exponer cómo realizar estas operaciones utilizando la sintaxis y el lenguaje de SQL, aprovecharemos también para explicar cómo llevar a cabo uniones (JOINs) de distintos dataframes utilizando herramientas propias de R.
A la hora de unir tablas o dataframes existen diversos aspectos que debemos tener en cuenta y dependerá de nuestros dataframes originales y del resultado que buscamos conseguir al realizar las uniones. Por ello existen diversos tipos de unión que, evidentemente, recibirán diferentes nombres. Intentaremos a lo largo del post abordar diferentes posibilidades utilizando información de algunos países procedentes del dataset gapminder del paquete del mismo nombre.
El paquete gapminder ofrece varios dataframes referentes a un conjunto importante de países. El dataframe filtrado recibe el nombre de gapminder:
gapminder::gapminder
## # A tibble: 1,704 x 6
## country continent year lifeExp pop gdpPercap
## <fct> <fct> <int> <dbl> <int> <dbl>
## 1 Afghanistan Asia 1952 28.8 8425333 779.
## 2 Afghanistan Asia 1957 30.3 9240934 821.
## 3 Afghanistan Asia 1962 32.0 10267083 853.
## 4 Afghanistan Asia 1967 34.0 11537966 836.
## 5 Afghanistan Asia 1972 36.1 13079460 740.
## 6 Afghanistan Asia 1977 38.4 14880372 786.
## 7 Afghanistan Asia 1982 39.9 12881816 978.
## 8 Afghanistan Asia 1987 40.8 13867957 852.
## 9 Afghanistan Asia 1992 41.7 16317921 649.
## 10 Afghanistan Asia 1997 41.8 22227415 635.
## # ... with 1,694 more rows
Un segundo dataset (continent_colors) contiene los colores que han asignado a cada continente:
gapminder::continent_colors
## Africa Americas Asia Europe Oceania
## "#7F3B08" "#A50026" "#40004B" "#276419" "#313695"
Un tercer dataset (country_codes) contiene información sobre los códigos de los países
gapminder::country_codes
## # A tibble: 187 x 3
## country iso_alpha iso_num
## <chr> <chr> <int>
## 1 Afghanistan AFG 4
## 2 Albania ALB 8
## 3 Algeria DZA 12
## 4 Angola AGO 24
## 5 Argentina ARG 32
## 6 Armenia ARM 51
## 7 Aruba ABW 533
## 8 Australia AUS 36
## 9 Austria AUT 40
## 10 Azerbaijan AZE 31
## # ... with 177 more rows
Y un cuarto dataframe (gapminder_unfiltered) contiene una mayor información que el dataset gapminder. Este data frame sin filtrar está compuesto por 3313 observaciones mientras que el paquete gapminder se conforma por 1704.
gapminder::gapminder_unfiltered
## # A tibble: 3,313 x 6
## country continent year lifeExp pop gdpPercap
## <fct> <fct> <int> <dbl> <int> <dbl>
## 1 Afghanistan Asia 1952 28.8 8425333 779.
## 2 Afghanistan Asia 1957 30.3 9240934 821.
## 3 Afghanistan Asia 1962 32.0 10267083 853.
## 4 Afghanistan Asia 1967 34.0 11537966 836.
## 5 Afghanistan Asia 1972 36.1 13079460 740.
## 6 Afghanistan Asia 1977 38.4 14880372 786.
## 7 Afghanistan Asia 1982 39.9 12881816 978.
## 8 Afghanistan Asia 1987 40.8 13867957 852.
## 9 Afghanistan Asia 1992 41.7 16317921 649.
## 10 Afghanistan Asia 1997 41.8 22227415 635.
## # ... with 3,303 more rows
En este post no vamos a utilizar la totalidad de los dataframes arriba expuestos. Como el objetivo de esta entrada consiste en explicar las distintas modalidades, o JOINs, y exponer su forma de llevarlas a cabo en SQL y en R, construiremos distintos subsets, partiendo de la información existente en los dataframes de {gapminder}, esperando que nuestros subsets nos permitan ejemplificar los distintos ejercicios de una forma más clara.
INNER JOINs
Supongamos que tenemos dos datasets, el primero con información sobre el PIB per cápita de una serie de países y el segundo con información sobre la esperanza de vida. No obstante los países del primer dataset (LEFT TABLE) y el segundo (RIGHT TABLE) no coinciden, habiendo países que están en ambos datasets pero otros países que se encuentran en uno de los datasets pero no en el otro. En el caso de que queramos generar un dataset donde tengamos información tanto del PIB per cápita como de la esperanza de vida, pero únicamente incluyendo los países que están en ambos datasets, utilizaremos los INNER JOINs.
El planteamiento general del INNER JOIN sería el que se observa en la figura siguiente. Vemos que el dataframe final solo incluye a los cuatros países presentes en ambas tablas (Australia, Canadá, Japón y España), excluyendo al resto de países, y la información de la segunda tabla (RIGHT TABLE) se une a la información presente en la primera (LEFT TABLE). Nótese que al utilizar los INNER JOINs conviene tener mucho cuidado para no perder información de forma indeseada.
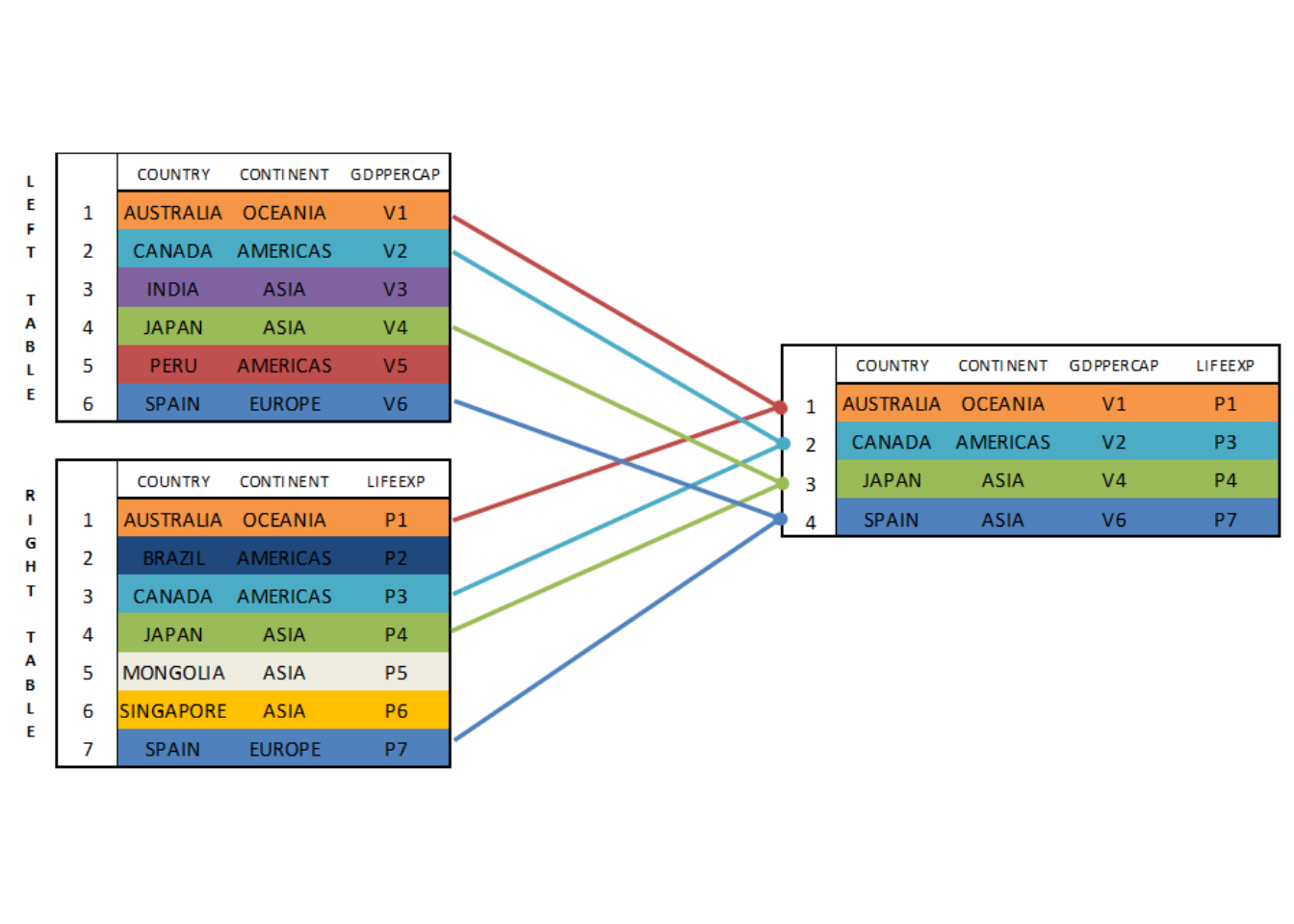
Aunque el anterior es el esquema general de los INNER JOINs, existen algunos aspectos que debemos tener en cuenta. Veámoslo con una serie de ejemplos. Para ello, en primer lugar vamos a realizar dos subsets que incluyan los mismos países de los expuestos en el dibujo previo, uno con información del PIB per cápita y otro con información de la esperanza de vida de dichos países.
# LEFT TABLE
vab_pc <- gapminder %>%
filter(year == 2007) %>%
filter(country %in% c("Spain", "Peru", "India", "Australia", "Japan", "Canada")) %>%
select(country, continent, gdpPercap)
# RIGHT TABLE
life_exp <- gapminder %>%
filter(year == 2007) %>%
filter(country %in% c("Spain", "Australia", "Singapore", "Canada", "Japan", "Mongolia", "Brazil")) %>%
select(country, continent, lifeExp)
Examinemos ambos datasets:
vab_pc
## # A tibble: 6 x 3
## country continent gdpPercap
## <fct> <fct> <dbl>
## 1 Australia Oceania 34435.
## 2 Canada Americas 36319.
## 3 India Asia 2452.
## 4 Japan Asia 31656.
## 5 Peru Americas 7409.
## 6 Spain Europe 28821.
life_exp
## # A tibble: 7 x 3
## country continent lifeExp
## <fct> <fct> <dbl>
## 1 Australia Oceania 81.2
## 2 Brazil Americas 72.4
## 3 Canada Americas 80.7
## 4 Japan Asia 82.6
## 5 Mongolia Asia 66.8
## 6 Singapore Asia 80.0
## 7 Spain Europe 80.9
Si queremos unir íntegramente el RIGHT TABLE al LEFT TABLE podemos indicar lo siguiente:
sqldf('SELECT *
FROM vab_pc
INNER JOIN life_exp
ON vab_pc.country = life_exp.country')
## country continent gdpPercap country continent lifeExp
## 1 Australia Oceania 34435.37 Australia Oceania 81.235
## 2 Canada Americas 36319.24 Canada Americas 80.653
## 3 Japan Asia 31656.07 Japan Asia 82.603
## 4 Spain Europe 28821.06 Spain Europe 80.941
En el caso anterior un dataset se ha unido al anterior incluyendo únicamente los países que coinciden en ambos. No obstante, el resultado final incluye columnas repetidas que no nos interesan (las columnas country y continent). Para conseguir un dataframe que se asemeje al del dibujo presentado arriba, formado por cuatro columnas únicamente, debemos realizar un serie de modificaciones, indicando que queremos solo cuatro columnas donde las dos primeras, al estar repetidas, serán las pertenecientes al primer dataset (p1),
sqldf('SELECT p1.country, p1.continent, gdpPercap, lifeExp
FROM vab_pc AS p1
INNER JOIN life_exp AS p2
ON p1.country = p2.country')
## country continent gdpPercap lifeExp
## 1 Australia Oceania 34435.37 81.235
## 2 Canada Americas 36319.24 80.653
## 3 Japan Asia 31656.07 82.603
## 4 Spain Europe 28821.06 80.941
Puede que queramos unir más de dataframe en un solo comando. Pongamos por ejemplo que queramos incluir los códigos de cada país, que como vimos están presentes en un dataframe denominado country_codes. Para unir los tres dataframes, situando los códigos en la primera columna del nuevo dataset, no es necesario realizar primero una unión y luego la segunda sino que podemos realizar la operación conjuntamente indicando todas los pasos en una sola expresión.
Para ello, en primer lugar guardamos el nuevo dataframe con el nombre codes.
codes <- gapminder::country_codes
Y realizamos una operación similar a la realizada previamente pero estableciendo dos INNER JOINs diferentes.
sqldf('SELECT iso_alpha AS code, p1.country, p1.continent, gdpPercap, lifeExp
FROM vab_pc AS p1
INNER JOIN life_exp AS p2
ON p1.country= p2.country
INNER JOIN codes AS p3
ON p1.country = p3.country')
## code country continent gdpPercap lifeExp
## 1 AUS Australia Oceania 34435.37 81.235
## 2 CAN Canada Americas 36319.24 80.653
## 3 JPN Japan Asia 31656.07 82.603
## 4 ESP Spain Europe 28821.06 80.941
Como en nuestro caso el nombre de las columnas que utilizamos para la unión es el mismo, podemos, de forma alternativa, establecer USING seguido por el nombre de la variable entre paréntesis (en nuestro caso country) para una mayor sencillez a la hora de redactar el código. El resultado, como vemos, es el mismo.
sqldf('SELECT iso_alpha AS code, p1.country, p1.continent, gdpPercap, lifeExp
FROM vab_pc AS p1
INNER JOIN life_exp AS p2
USING (country)
INNER JOIN codes AS p3
USING (country)')
## code country continent gdpPercap lifeExp
## 1 AUS Australia Oceania 34435.37 81.235
## 2 CAN Canada Americas 36319.24 80.653
## 3 JPN Japan Asia 31656.07 82.603
## 4 ESP Spain Europe 28821.06 80.941
SELF-JOINs
Se denominan SELF-JOINs cuando las uniones las realizamos utilizando el mismo dataframe de origen. Es decir, la tabla final, en lugar de ser resultado de dos tablas distintas es resultado de una sola. Aunque parezca extraño en el día a día este tipo de operaciones pueden ser, como veremos en el ejemplo, muy habituales. En nuestro ejemplo particular, utilizando datos del dataframe gapminder(), supongamos que nos interesa generar un dataset donde quede establecido en una columna la esperanza de vida en el año inicial del periodo (1952), en otra columna la esperanza de vida en el año final (2007) y en una tercera columna el crecimiento entre 1952 y 2007. El objetivo de este tipo de análisis consiste en determinar el incremento o descenso de la esperanza de vida por país durante ese periodo.
sqldf('SELECT p1.country,
p1.lifeExp AS lifeExp1952,
p2.lifeExp AS lifeExp2007,
((p2.lifeExp - p1.lifeExp)/p1.lifeExp) *100 AS crecimiento
FROM gapminder AS p1
INNER JOIN gapminder AS p2
ON p1.country = p2.country
AND p1.year = p2.year-55
LIMIT 10')
## country lifeExp1952 lifeExp2007 crecimiento
## 1 Afghanistan 28.801 43.828 52.17527
## 2 Albania 55.230 76.423 38.37226
## 3 Algeria 43.077 72.301 67.84131
## 4 Angola 30.015 42.731 42.36548
## 5 Argentina 62.485 75.320 20.54093
## 6 Australia 69.120 81.235 17.52749
## 7 Austria 66.800 79.829 19.50449
## 8 Bahrain 50.939 75.635 48.48152
## 9 Bangladesh 37.484 64.062 70.90492
## 10 Belgium 68.000 79.441 16.82500
En R podemos obtener el mismo resultado utilizando la función filter(), para seleccionar el año inicial y final, junto a la función spread() para generar las dos columnas, una para 1952 y otra para 2007, y la función mutate() para crear una nueva columna que indique el crecimiento de la siguiente forma:
gapminder %>%
filter(year == c(1952,2007)) %>%
select(country, continent, year, lifeExp) %>%
spread(key= year, lifeExp) %>%
mutate(crecimiento = ((`2007`-`1952`)/`1952`)*100) %>%
head(n=10)
## # A tibble: 10 x 5
## country continent `1952` `2007` crecimiento
## <fct> <fct> <dbl> <dbl> <dbl>
## 1 Afghanistan Asia 28.8 43.8 52.2
## 2 Albania Europe 55.2 76.4 38.4
## 3 Algeria Africa 43.1 72.3 67.8
## 4 Angola Africa 30.0 42.7 42.4
## 5 Argentina Americas 62.5 75.3 20.5
## 6 Australia Oceania 69.1 81.2 17.5
## 7 Austria Europe 66.8 79.8 19.5
## 8 Bahrain Asia 50.9 75.6 48.5
## 9 Bangladesh Asia 37.5 64.1 70.9
## 10 Belgium Europe 68 79.4 16.8
LEFT JOINs y RIGHT JOINs
En oposición a los INNER JOINs, los LEFT JOINs, los RIGHT JOINs y los FULL JOINs (que veremos posteriormente) se conocen como OUTER JOINs. Estos últimos, aunque con diferencias, conservan las observaciones que aparecen, al menos, en una de las dos tablas originales.
LEFT JOINs
Los LEFT JOINs y los RIGHT JOINs responden a la misma lógica, aunque los primeros suelen ser más utilizados que los segundos. Supongamos de nuevo que tenemos las dos tablas del gráfico presentado previamente. En esta ocasión nos interesa generar una nueva tabla partiendo de la información presente en las otras dos, pero en este caso necesitamos que la tabla nueva contenga todas las variables presentes en la primera tabla (LEFT TABLE). En la práctica los LEFT JOINs son los más utilizados porque permiten añadir información proveniente de otra tabla pero conservando las observaciones del dataframe sobre las que se está trabajando.
En nuestro caso, al realizar un LEFT JOIN, la tabla generada incluirá Australia, Canadá, India, Japón, Perú y España. No obstante, como en la segunda tabla no hay información de India y Perú, la nueva tabla tendrá missing values para dichos países en relación a la esperanza de vida. En la figura siguiente se observa la dinámica descrita de los LEFT JOINs.
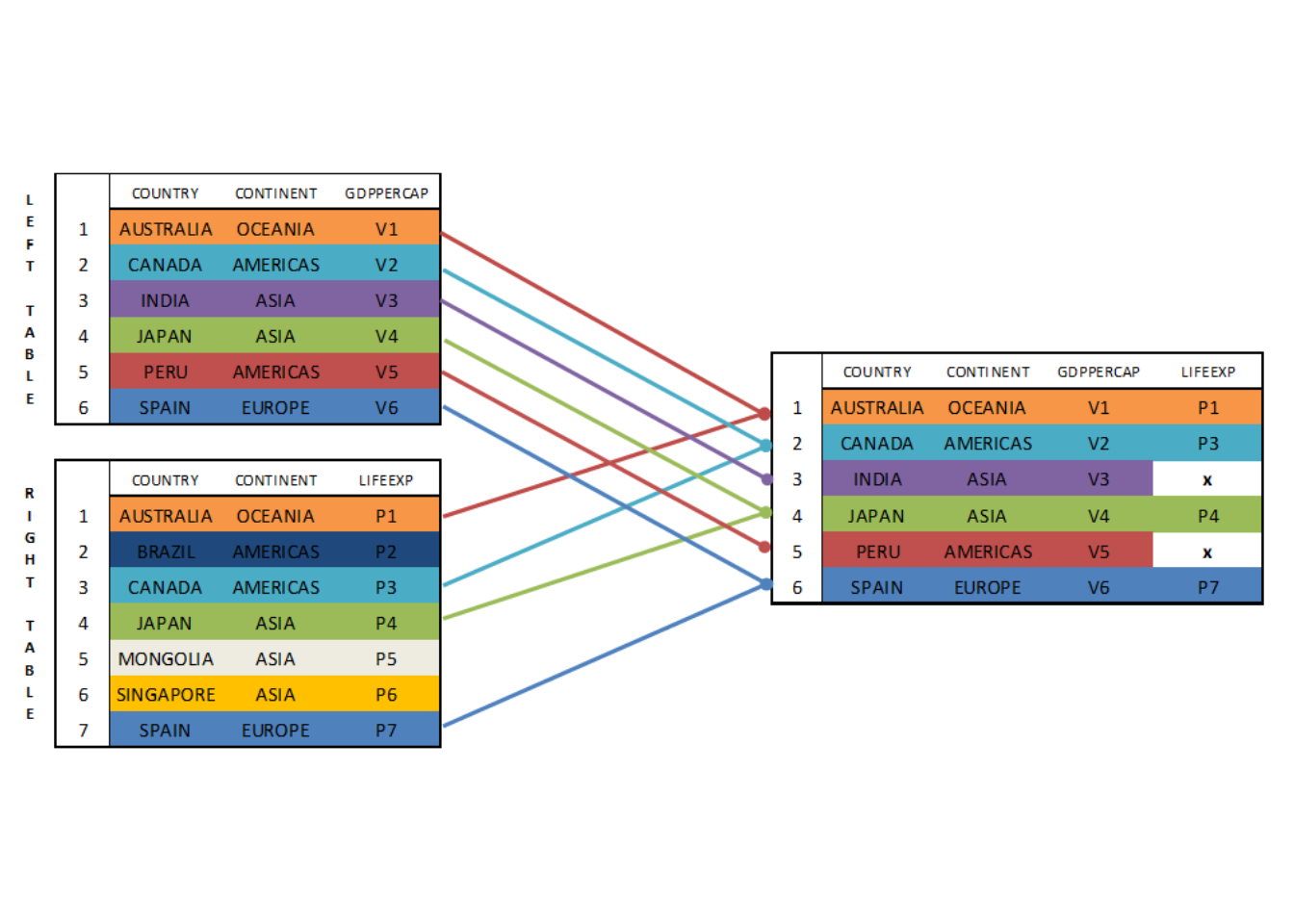
En sqldf podemos realizar el LEFT JOIN modificando ligeramente la expresión del INNER JOIN expuesta en el apartado previo.
sqldf('SELECT p1.country, p1.continent, gdpPercap, lifeExp
FROM vab_pc AS p1
LEFT JOIN life_exp AS p2
ON p1.country = p2.country')
## country continent gdpPercap lifeExp
## 1 Australia Oceania 34435.367 81.235
## 2 Canada Americas 36319.235 80.653
## 3 India Asia 2452.210 NA
## 4 Japan Asia 31656.068 82.603
## 5 Peru Americas 7408.906 NA
## 6 Spain Europe 28821.064 80.941
En R existen distintas posibilidades para realizar esta misma operación. Podemos, por un lado, utilizar la sintaxis base de R, y utilizar la función merge() para hacer el JOIN o, por otro lado, utilizar la función left_join() del paquete {dplyr}.
# BASE R:
merge(vab_pc, life_exp, all.x = TRUE)
## country continent gdpPercap lifeExp
## 1 Australia Oceania 34435.367 81.235
## 2 Canada Americas 36319.235 80.653
## 3 India Asia 2452.210 NA
## 4 Japan Asia 31656.068 82.603
## 5 Peru Americas 7408.906 NA
## 6 Spain Europe 28821.064 80.941
# DPLYR:
vab_pc %>%
left_join(life_exp, by = c("country", "continent"))
## # A tibble: 6 x 4
## country continent gdpPercap lifeExp
## <fct> <fct> <dbl> <dbl>
## 1 Australia Oceania 34435. 81.2
## 2 Canada Americas 36319. 80.7
## 3 India Asia 2452. NA
## 4 Japan Asia 31656. 82.6
## 5 Peru Americas 7409. NA
## 6 Spain Europe 28821. 80.9
RIGHT JOINs
Por su parte, si queremos conservar todas las observaciones del RIGHT TABLE, completando dicha tabla con los valores del LEFT table haremos uso del RIGHT JOIN. En este caso, como se refleja en la figura siguiente, la tabla final se compondría de los siete países del RIGHT TABLE (Australia, Brasil, Canadá, Japón, Mongolia, Singapur y España), aunque en tres de ellos, en aquellos países que no se encuentran en el LEFT TABLE, faltarán los valores del PIB por habitante.
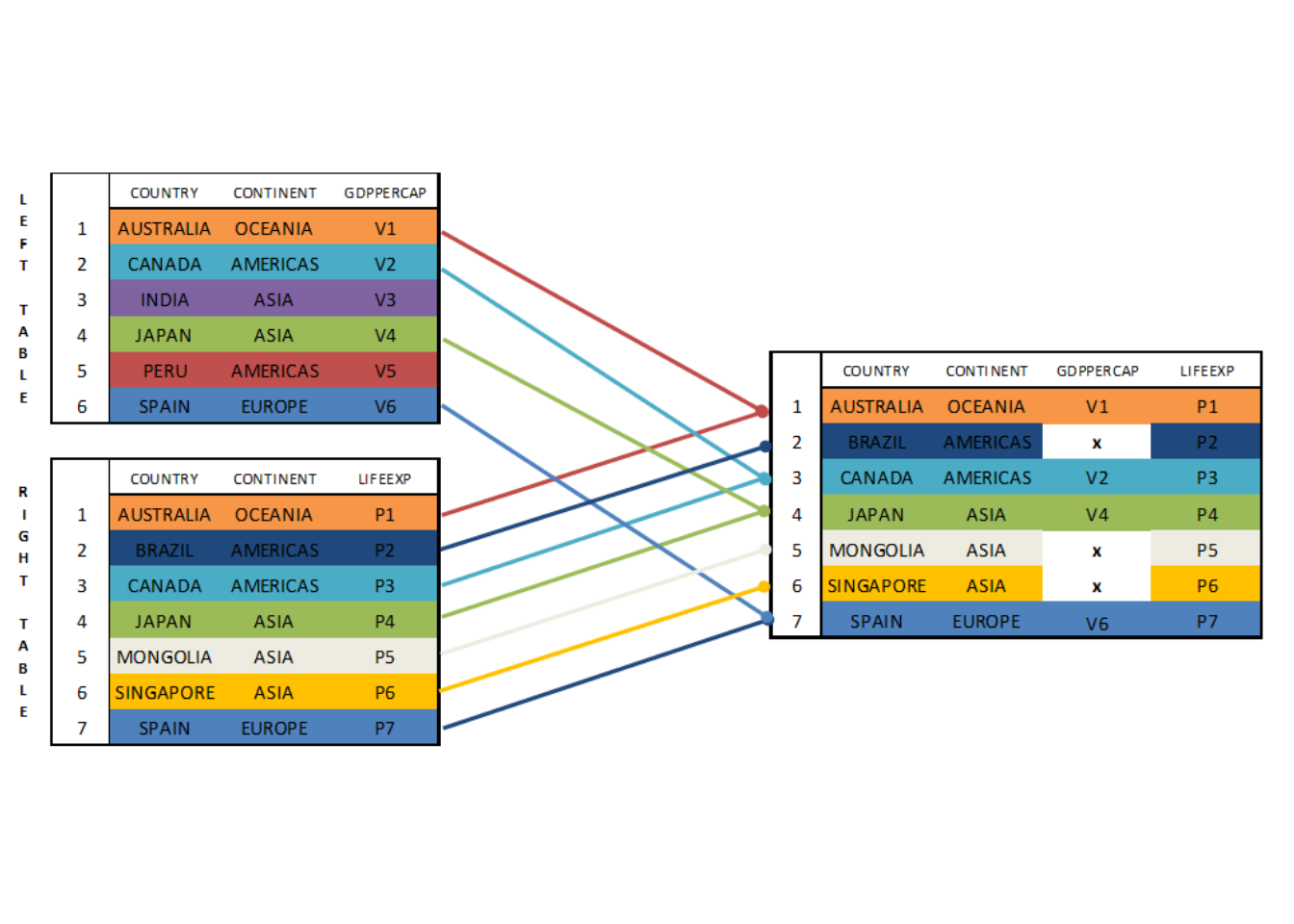
En el caso anterior hemos establecido las columnas a utilizar para realizar el JOIN con la expresión by= c("country", "continent"). En general la función utiliza para la unión todas las variables que aparecen en ambas tablas (by= NULL), por lo que en este caso en particular no habría sido necesario indicar las columnas a utilizar. En el caso de que las columnas tuvieran diferente nombre en cada dataset, pongamos por ejemplo que en nuestro caso los países aparecen como “name” y como “country” respectivamente, podríamos indicar (by= c("name" = "country")).
Por desgracia el paquete {sqldf} no permite realizar RIGHT JOINs. No obstante, para no dejar la explicación un poco “coja” vamos a mostrar cómo realizar estas uniones con R, que evidentemente se asemejará a los procedimientos utilizados al realizar LEFT JOINs vistos arriba (Base R y con el paquete {dplyr}).
# BASE R:
merge(vab_pc, life_exp, all.y = TRUE)
## country continent gdpPercap lifeExp
## 1 Australia Oceania 34435.37 81.235
## 2 Brazil Americas NA 72.390
## 3 Canada Americas 36319.24 80.653
## 4 Japan Asia 31656.07 82.603
## 5 Mongolia Asia NA 66.803
## 6 Singapore Asia NA 79.972
## 7 Spain Europe 28821.06 80.941
# DPLYR:
vab_pc %>%
right_join(life_exp, by = c("country", "continent"))
## # A tibble: 7 x 4
## country continent gdpPercap lifeExp
## <fct> <fct> <dbl> <dbl>
## 1 Australia Oceania 34435. 81.2
## 2 Canada Americas 36319. 80.7
## 3 Japan Asia 31656. 82.6
## 4 Spain Europe 28821. 80.9
## 5 Brazil Americas NA 72.4
## 6 Mongolia Asia NA 66.8
## 7 Singapore Asia NA 80.0
FULL JOINs
Por último, cabe la posibilidad que al unir dos dataframes nos interese conservar las observaciones presentes en ambos. Es decir, cabe la posibilidad de que en el dataset que queremos generar nos interese que aparezcan todos los países del LEFT TABLE y también todos los países del RIGHT TABLE. Evidentemente, en nuestro caso particular, la tabla generada contendrá missing values tanto en la columna del VAB por habitante como en la columna de la esperanza de vida. El esquema de dicho proceso sería el siguiente:
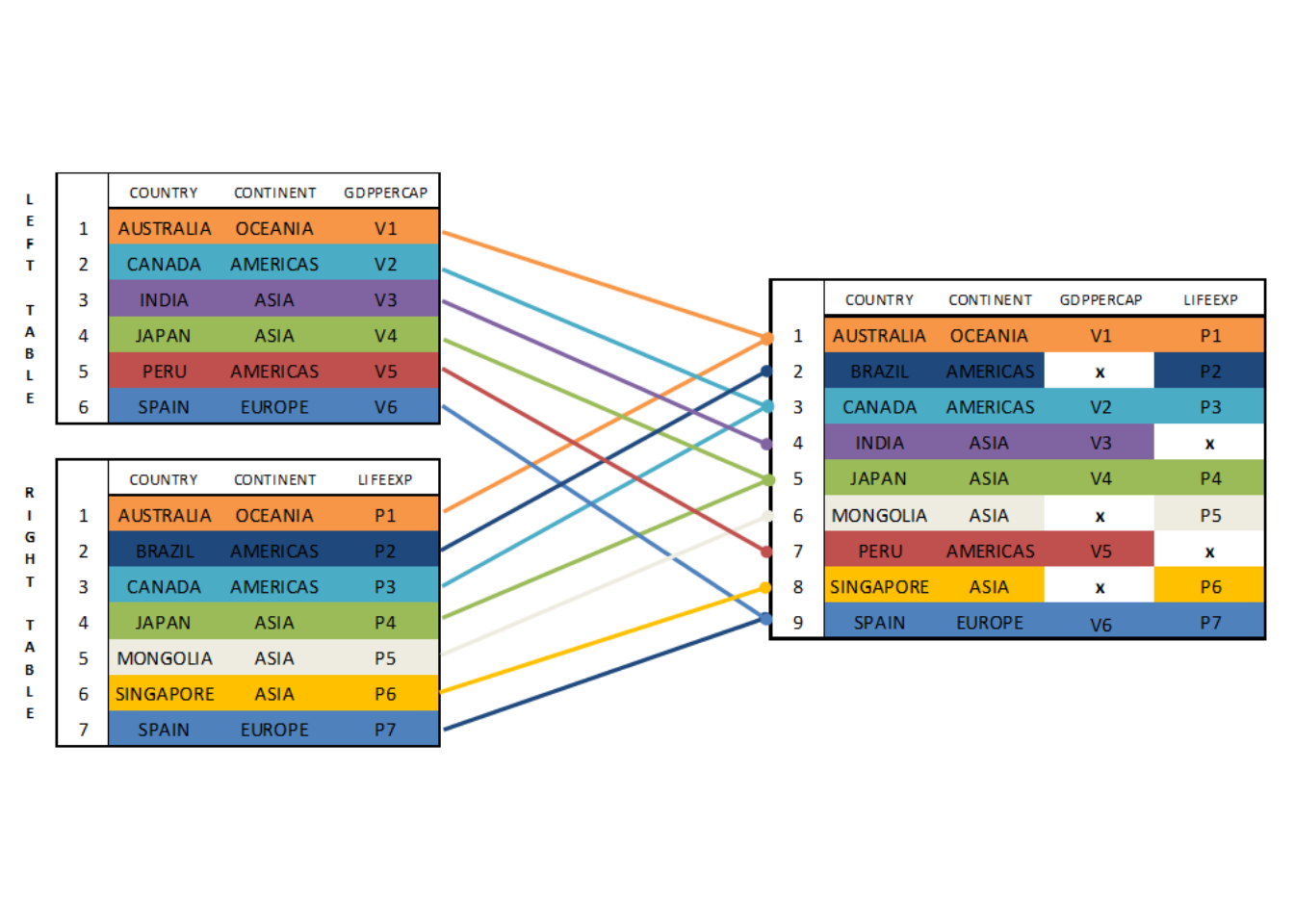
Al igual que sucedía con los RIGHT JOINs, el paquete {sqldf} no permite, al menos a día de hoy, realizar FULL JOINs. No obstante, de nuevo, con la finalidad de no dejar el post un tanto incompleto vamos a exponer cómo realizar dichas operaciones en R.
De nuevo existen diversas formas para realizar FULL JOINs en R. Podemos utilizar la sintaxis básica de R, utilizando la función merge() o las funciones de {dplyr}, de forma análoga a cómo hemos realizado los OUTER JOINS previos.
# BASE R:
merge(vab_pc, life_exp, all.x = TRUE, all.y = TRUE)
## country continent gdpPercap lifeExp
## 1 Australia Oceania 34435.367 81.235
## 2 Brazil Americas NA 72.390
## 3 Canada Americas 36319.235 80.653
## 4 India Asia 2452.210 NA
## 5 Japan Asia 31656.068 82.603
## 6 Mongolia Asia NA 66.803
## 7 Peru Americas 7408.906 NA
## 8 Singapore Asia NA 79.972
## 9 Spain Europe 28821.064 80.941
# DPLYR:
vab_pc %>%
full_join(life_exp, by = c("country", "continent"))
## # A tibble: 9 x 4
## country continent gdpPercap lifeExp
## <fct> <fct> <dbl> <dbl>
## 1 Australia Oceania 34435. 81.2
## 2 Canada Americas 36319. 80.7
## 3 India Asia 2452. NA
## 4 Japan Asia 31656. 82.6
## 5 Peru Americas 7409. NA
## 6 Spain Europe 28821. 80.9
## 7 Brazil Americas NA 72.4
## 8 Mongolia Asia NA 66.8
## 9 Singapore Asia NA 80.0
SEMI JOINs y ANTI JOINs
SEMI JOINs
Los SEMI JOINs nos permiten filtrar observaciones en lugar de variables. Pongamos que nos interesa filtrar el dataset vab_pc en función de si los países ahí incluidos están también en el dataset life_exp (o viceversa). Para ello debemos especificar las columnas del LEFT TABLE que nos interesan y especificar también una condición, donde se requiera que los nombres se encuentren también en una segunda tabla (RIGHT TABLE). Por consiguiente, el SEMI JOIN no añadiría ninguna nueva columna al dataset original (LEFT TABLE), únicamente identifica aquellos países que se encuentran en el segundo dataset.
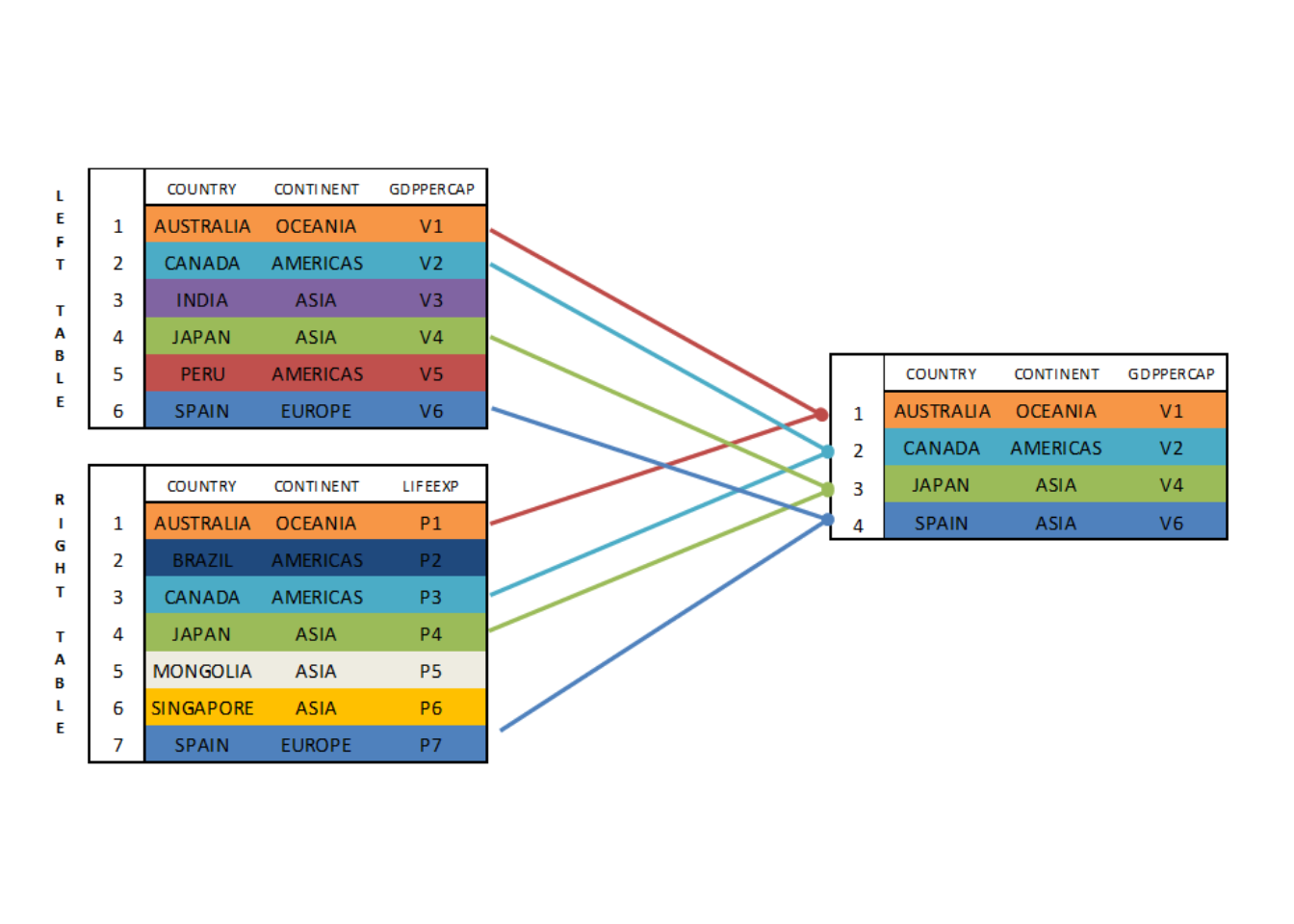
Realizamos el SEMI JOIN de la siguiente forma:
sqldf('SELECT country, continent, gdpPercap
FROM vab_pc
WHERE country IN
(SELECT country
FROM life_exp)')
## country continent gdpPercap
## 1 Australia Oceania 34435.37
## 2 Canada Americas 36319.24
## 3 Japan Asia 31656.07
## 4 Spain Europe 28821.06
sqldf('SELECT country, continent, lifeExp
FROM life_exp
WHERE country IN
(SELECT country
FROM vab_pc)')
## country continent lifeExp
## 1 Australia Oceania 81.235
## 2 Canada Americas 80.653
## 3 Japan Asia 82.603
## 4 Spain Europe 80.941
En R el comando sería:
semi_join(vab_pc, life_exp)
## Joining, by = c("country", "continent")
## # A tibble: 4 x 3
## country continent gdpPercap
## <fct> <fct> <dbl>
## 1 Australia Oceania 34435.
## 2 Canada Americas 36319.
## 3 Japan Asia 31656.
## 4 Spain Europe 28821.
semi_join(life_exp, vab_pc)
## Joining, by = c("country", "continent")
## # A tibble: 4 x 3
## country continent lifeExp
## <fct> <fct> <dbl>
## 1 Australia Oceania 81.2
## 2 Canada Americas 80.7
## 3 Japan Asia 82.6
## 4 Spain Europe 80.9
Nótese que es posible realizar una operación similar con %in% de la siguiente forma:
vab_pc %>%
filter(country %in% life_exp$country)
## # A tibble: 4 x 3
## country continent gdpPercap
## <fct> <fct> <dbl>
## 1 Australia Oceania 34435.
## 2 Canada Americas 36319.
## 3 Japan Asia 31656.
## 4 Spain Europe 28821.
ANTI JOINs
El ANTI JOIN es el mecanismo inverso al SEMI JOIN. Su finalidad consiste en identificar aquellas observaciones que no se encuentran en el segundo dataset. Aunque parezca de poca relevancia, los ANTI JOINS pueden resultar de gran utilidad a la hora de identificar errores o discrepancias entre dos datasets.
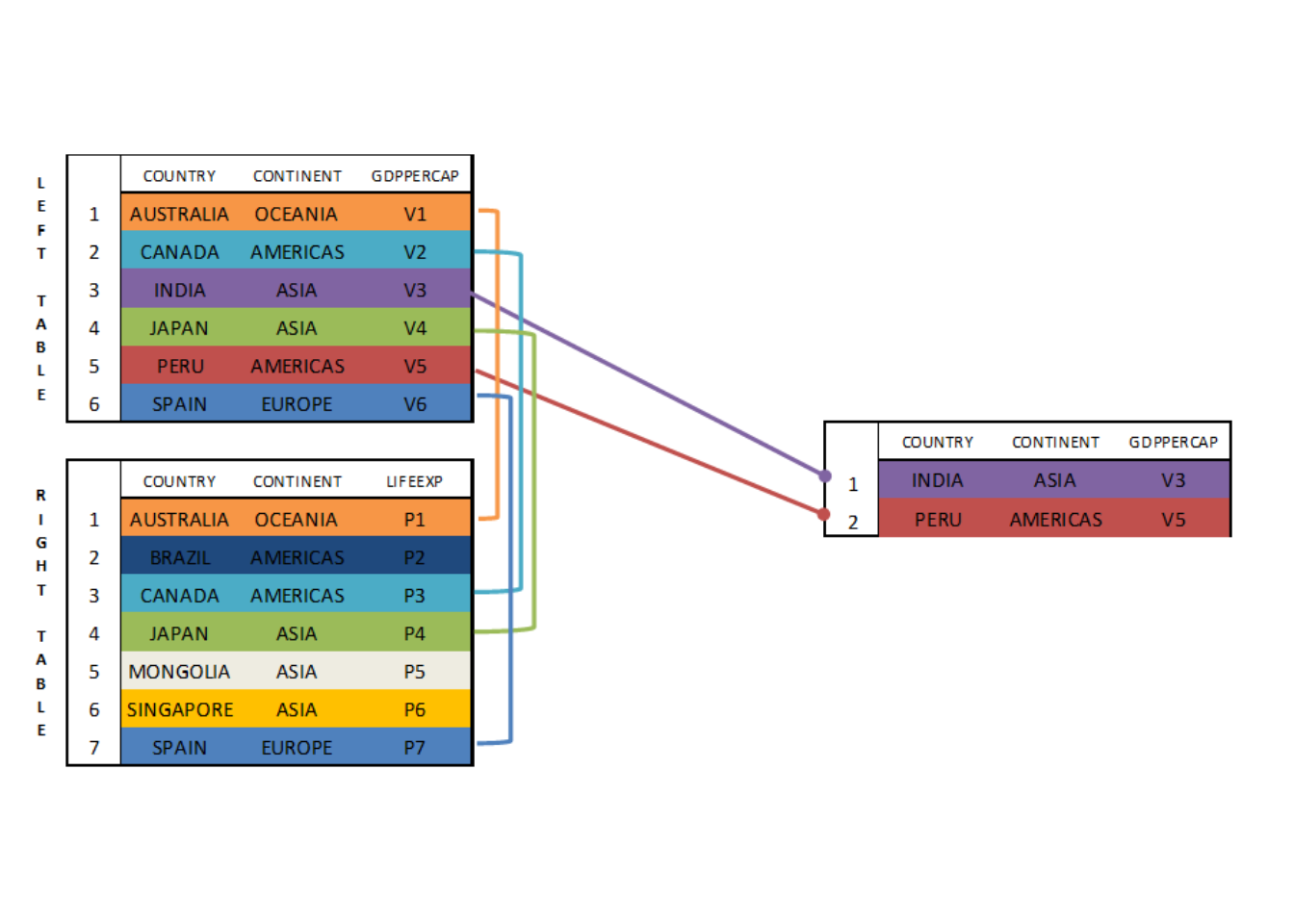
En nuestro caso los ANTI JOIN se realizarían de la siguiente forma:
sqldf('SELECT country, continent, gdpPercap
FROM vab_pc
WHERE country NOT IN
(SELECT country
FROM life_exp)')
## country continent gdpPercap
## 1 India Asia 2452.210
## 2 Peru Americas 7408.906
sqldf('SELECT country, continent, lifeExp
FROM life_exp
WHERE country NOT IN
(SELECT country
FROM vab_pc)')
## country continent lifeExp
## 1 Brazil Americas 72.390
## 2 Mongolia Asia 66.803
## 3 Singapore Asia 79.972
Y en R:
anti_join(vab_pc, life_exp)
## Joining, by = c("country", "continent")
## # A tibble: 2 x 3
## country continent gdpPercap
## <fct> <fct> <dbl>
## 1 India Asia 2452.
## 2 Peru Americas 7409.
anti_join(life_exp, vab_pc)
## Joining, by = c("country", "continent")
## # A tibble: 3 x 3
## country continent lifeExp
## <fct> <fct> <dbl>
## 1 Brazil Americas 72.4
## 2 Mongolia Asia 66.8
## 3 Singapore Asia 80.0
Alternativamente podemos utilizar %nin% del paquete {Hmisc} de forma similar al realizado previamente con %in%. Téngase en cuenta que %nin% sería un operador inverso a %in%.
vab_pc %>%
filter(country %nin% life_exp$country)
## # A tibble: 2 x 3
## country continent gdpPercap
## <fct> <fct> <dbl>
## 1 India Asia 2452.
## 2 Peru Americas 7409.
OTRO TIPO DE JOINS
Existen algunos otros operadores que nos pueden resultar de utilidad a la hora de realizar nuestros análisis, sobre todo cuando estamos realizando operaciones con dos datasets que presentan las mismas variables.
Para ejemplificar la operatividad de estas funciones vamos a realizar una pequeña variación en nuestros datasets (LEFT TABLE Y RIGHT TABLE). En este caso ambos datasets presentan información sobre el PIB per cápita y sobe la esperanza de vida aunque se diferenciarán en los países que componen cada uno de ellos.
A <- gapminder %>%
filter(year == 2007) %>%
filter(country %in% c("Spain", "Peru", "India", "Australia", "Japan", "Canada")) %>%
select(country, continent, gdpPercap, lifeExp)
B <- gapminder %>%
filter(year == 2007) %>%
filter(country %in% c("Spain", "Australia", "Singapore", "Canada", "Japan", "Mongolia", "Brazil")) %>%
select(country, continent, gdpPercap, lifeExp)
A
## # A tibble: 6 x 4
## country continent gdpPercap lifeExp
## <fct> <fct> <dbl> <dbl>
## 1 Australia Oceania 34435. 81.2
## 2 Canada Americas 36319. 80.7
## 3 India Asia 2452. 64.7
## 4 Japan Asia 31656. 82.6
## 5 Peru Americas 7409. 71.4
## 6 Spain Europe 28821. 80.9
B
## # A tibble: 7 x 4
## country continent gdpPercap lifeExp
## <fct> <fct> <dbl> <dbl>
## 1 Australia Oceania 34435. 81.2
## 2 Brazil Americas 9066. 72.4
## 3 Canada Americas 36319. 80.7
## 4 Japan Asia 31656. 82.6
## 5 Mongolia Asia 3096. 66.8
## 6 Singapore Asia 47143. 80.0
## 7 Spain Europe 28821. 80.9
INTERSECT
La función INTERSECT devuelve aquellas observaciones que se encuentren en ambos datasets. Esta función se asemeja al INNER JOIN pero, recordemos, en este caso se requiere que ambos datasets estén conformados por las mismas columnas tal y como se observa en la figura siguiente.
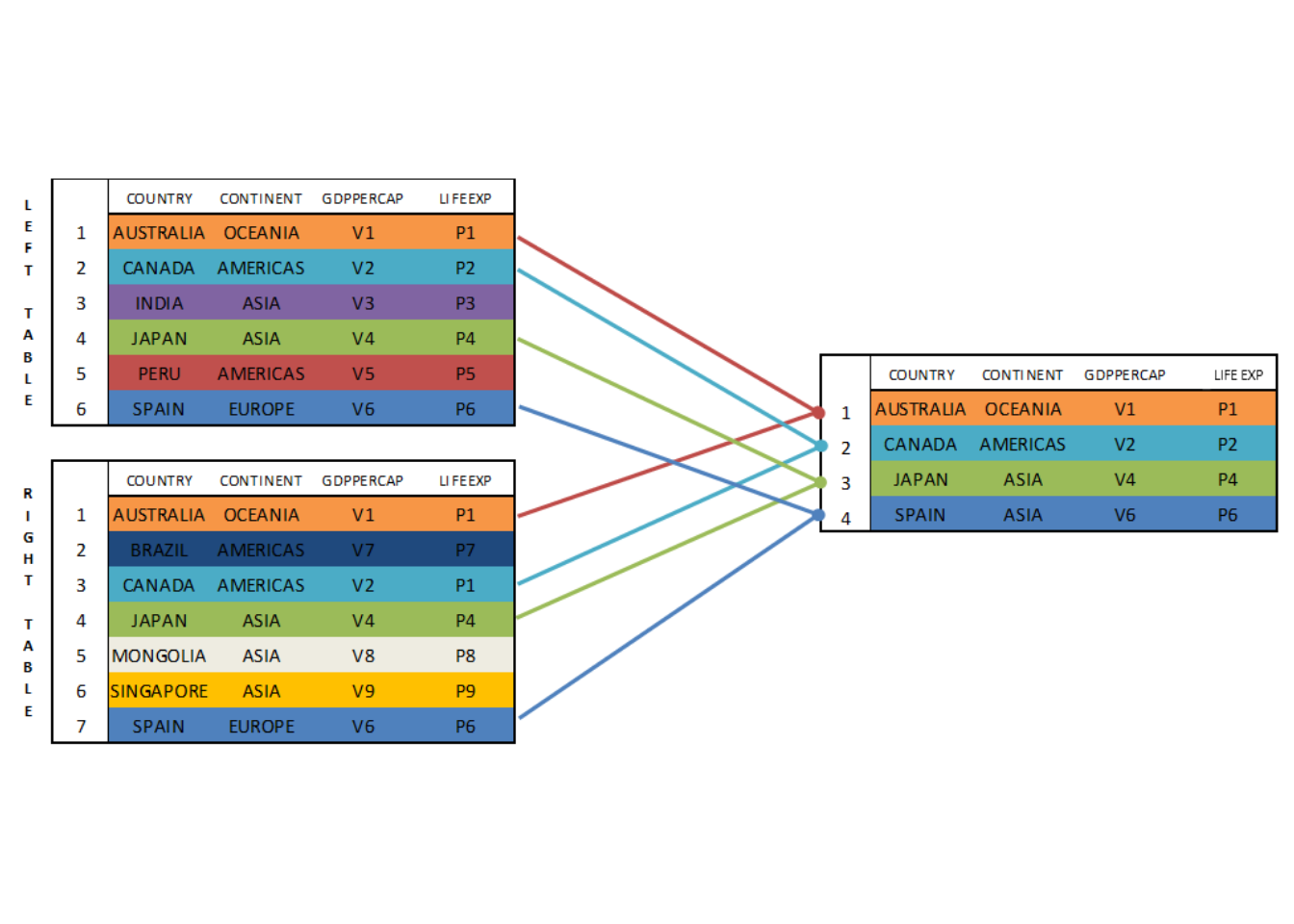
Su realización en SQL sería:
sqldf('SELECT country, continent, gdpPercap
FROM A
INTERSECT
SELECT country, continent, gdpPercap
FROM B')
## country continent gdpPercap
## 1 Australia Oceania 34435.37
## 2 Canada Americas 36319.24
## 3 Japan Asia 31656.07
## 4 Spain Europe 28821.06
En R utilizamos la función intersect() para obtener el resultado anterior.
intersect(A,B)
## # A tibble: 4 x 4
## country continent gdpPercap lifeExp
## <fct> <fct> <dbl> <dbl>
## 1 Australia Oceania 34435. 81.2
## 2 Canada Americas 36319. 80.7
## 3 Japan Asia 31656. 82.6
## 4 Spain Europe 28821. 80.9
UNION Y UNION ALL
La función UNION añade al dataset las observaciones del RIGTH TABLE no presentes en el LEFT TABLE. En nuestro caso podemos comprobar que Brasil, Mongolia y Singapur han sido añadidos al LEFT TABLE original.
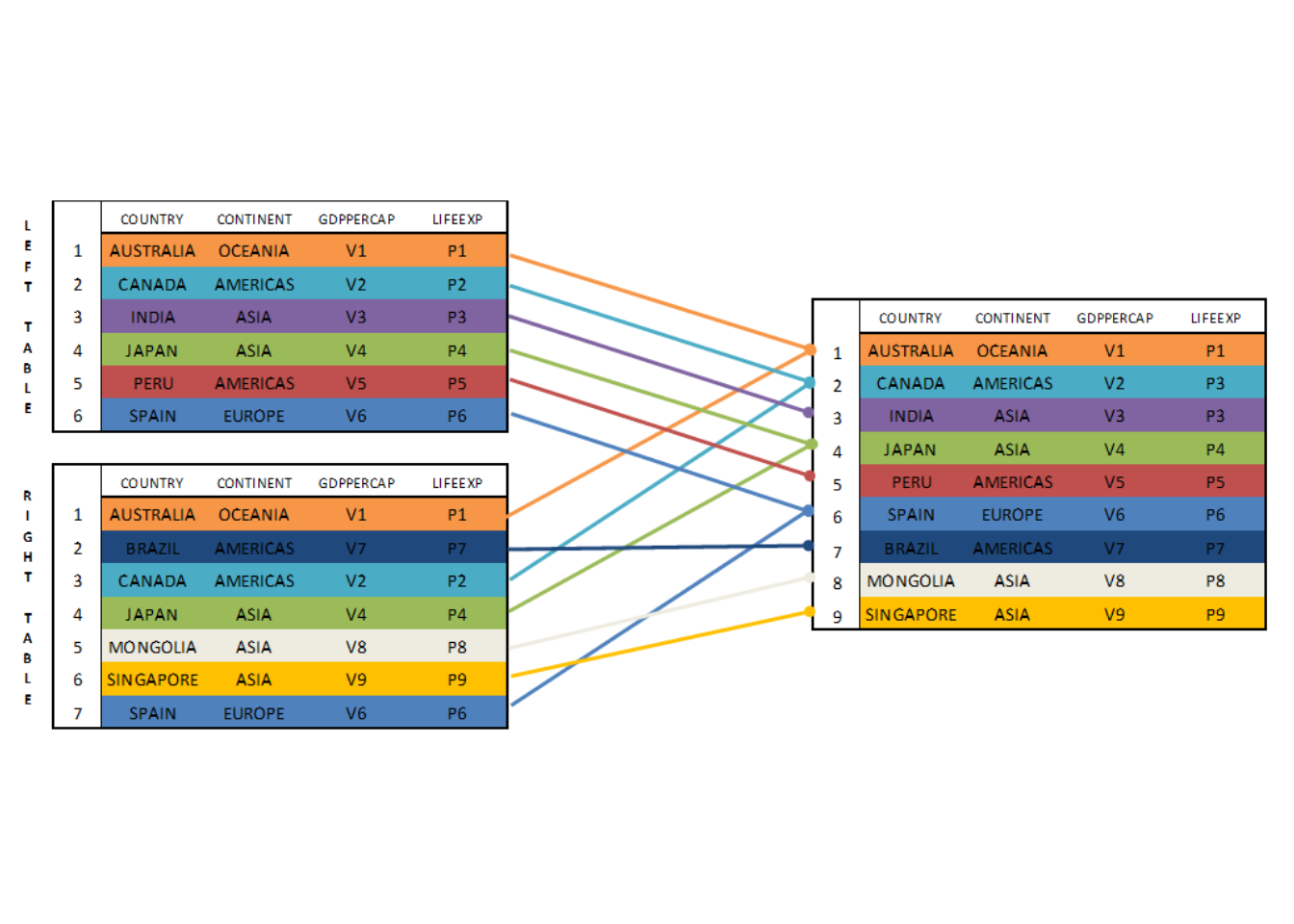
Por su parte, UNION ALL realiza la misma operación que UNION pero, a diferencia de esta, UNION ALL devuelve todas las observaciones de ambos dataframes, pero repitiendo las observaciones que se encuentren en los dos. Por consiguiente, en nuestro caso particular Australia, Canadá, Japón y España aparecerán dos veces en el dataset final.
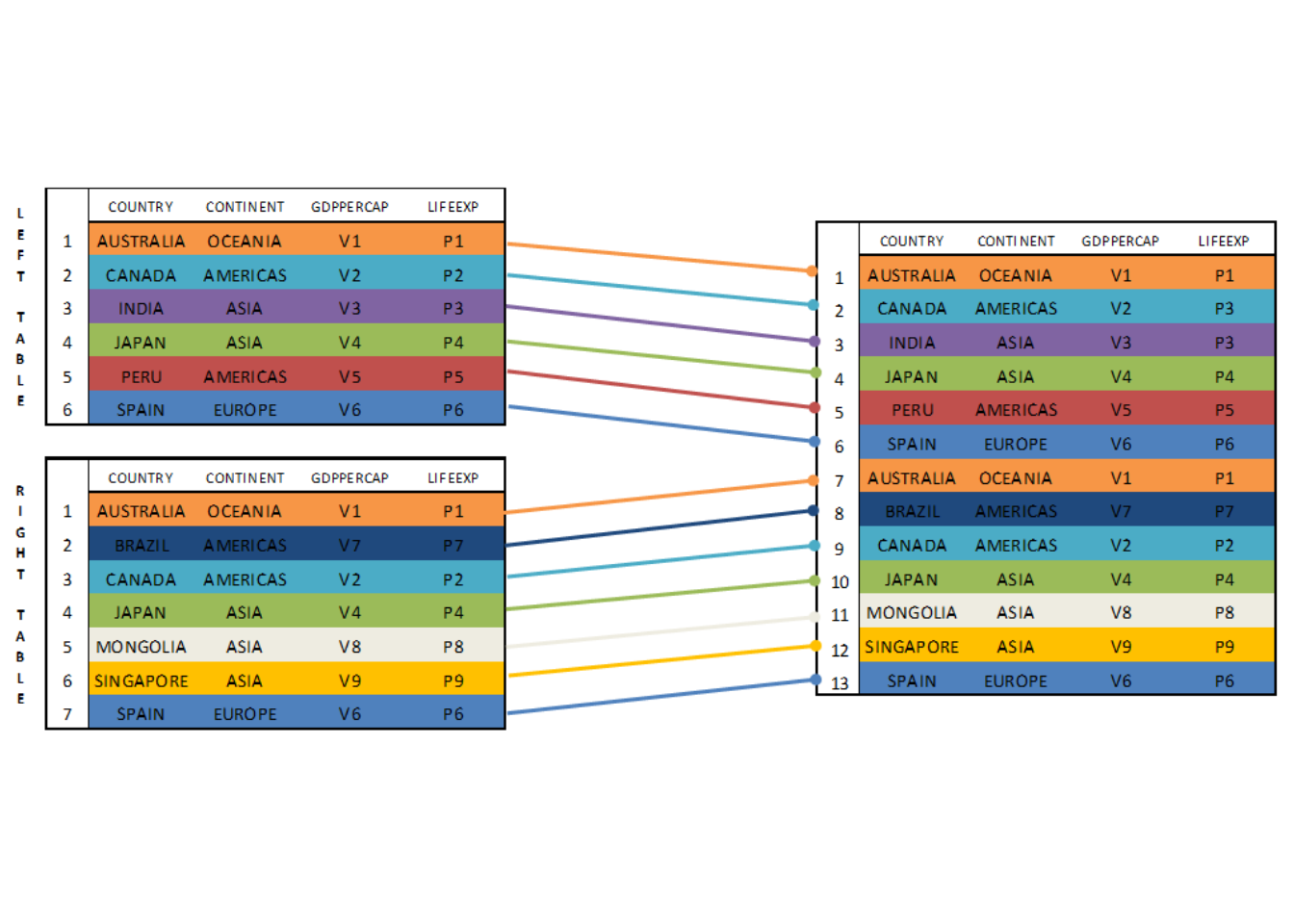
# UNION
sqldf('SELECT country, continent, gdpPercap
FROM A
UNION
SELECT country, continent, gdpPercap
FROM B')
## country continent gdpPercap
## 1 Australia Oceania 34435.367
## 2 Brazil Americas 9065.801
## 3 Canada Americas 36319.235
## 4 India Asia 2452.210
## 5 Japan Asia 31656.068
## 6 Mongolia Asia 3095.772
## 7 Peru Americas 7408.906
## 8 Singapore Asia 47143.180
## 9 Spain Europe 28821.064
# UNION ALL
sqldf('SELECT country, continent, gdpPercap
FROM A
UNION ALL
SELECT country, continent, gdpPercap
FROM B')
## country continent gdpPercap
## 1 Australia Oceania 34435.367
## 2 Canada Americas 36319.235
## 3 India Asia 2452.210
## 4 Japan Asia 31656.068
## 5 Peru Americas 7408.906
## 6 Spain Europe 28821.064
## 7 Australia Oceania 34435.367
## 8 Brazil Americas 9065.801
## 9 Canada Americas 36319.235
## 10 Japan Asia 31656.068
## 11 Mongolia Asia 3095.772
## 12 Singapore Asia 47143.180
## 13 Spain Europe 28821.064
El nombre de los operadores en R es igual, union() y union_all(), aunque la operatividad resulta algo más sencilla:
union (A,B)
## # A tibble: 9 x 4
## country continent gdpPercap lifeExp
## <fct> <fct> <dbl> <dbl>
## 1 Australia Oceania 34435. 81.2
## 2 Canada Americas 36319. 80.7
## 3 India Asia 2452. 64.7
## 4 Japan Asia 31656. 82.6
## 5 Peru Americas 7409. 71.4
## 6 Spain Europe 28821. 80.9
## 7 Brazil Americas 9066. 72.4
## 8 Mongolia Asia 3096. 66.8
## 9 Singapore Asia 47143. 80.0
union_all(A,B)
## # A tibble: 13 x 4
## country continent gdpPercap lifeExp
## <fct> <fct> <dbl> <dbl>
## 1 Australia Oceania 34435. 81.2
## 2 Canada Americas 36319. 80.7
## 3 India Asia 2452. 64.7
## 4 Japan Asia 31656. 82.6
## 5 Peru Americas 7409. 71.4
## 6 Spain Europe 28821. 80.9
## 7 Australia Oceania 34435. 81.2
## 8 Brazil Americas 9066. 72.4
## 9 Canada Americas 36319. 80.7
## 10 Japan Asia 31656. 82.6
## 11 Mongolia Asia 3096. 66.8
## 12 Singapore Asia 47143. 80.0
## 13 Spain Europe 28821. 80.9
EXCEPT
EXCEPT devuelve las observaciones presentes en el LEFT TABLE pero no en el RIGHT TABLE. Por consiguiente, en nuestro caso en particular, EXCEPT devolverá los países India y Perú, que se encuentran en A pero no en B. Si cambiásemos el orden en la expresión, de (A,B) a (B,A) EXCEPT nos devolvería Brasil, Mongolia y Singapur.
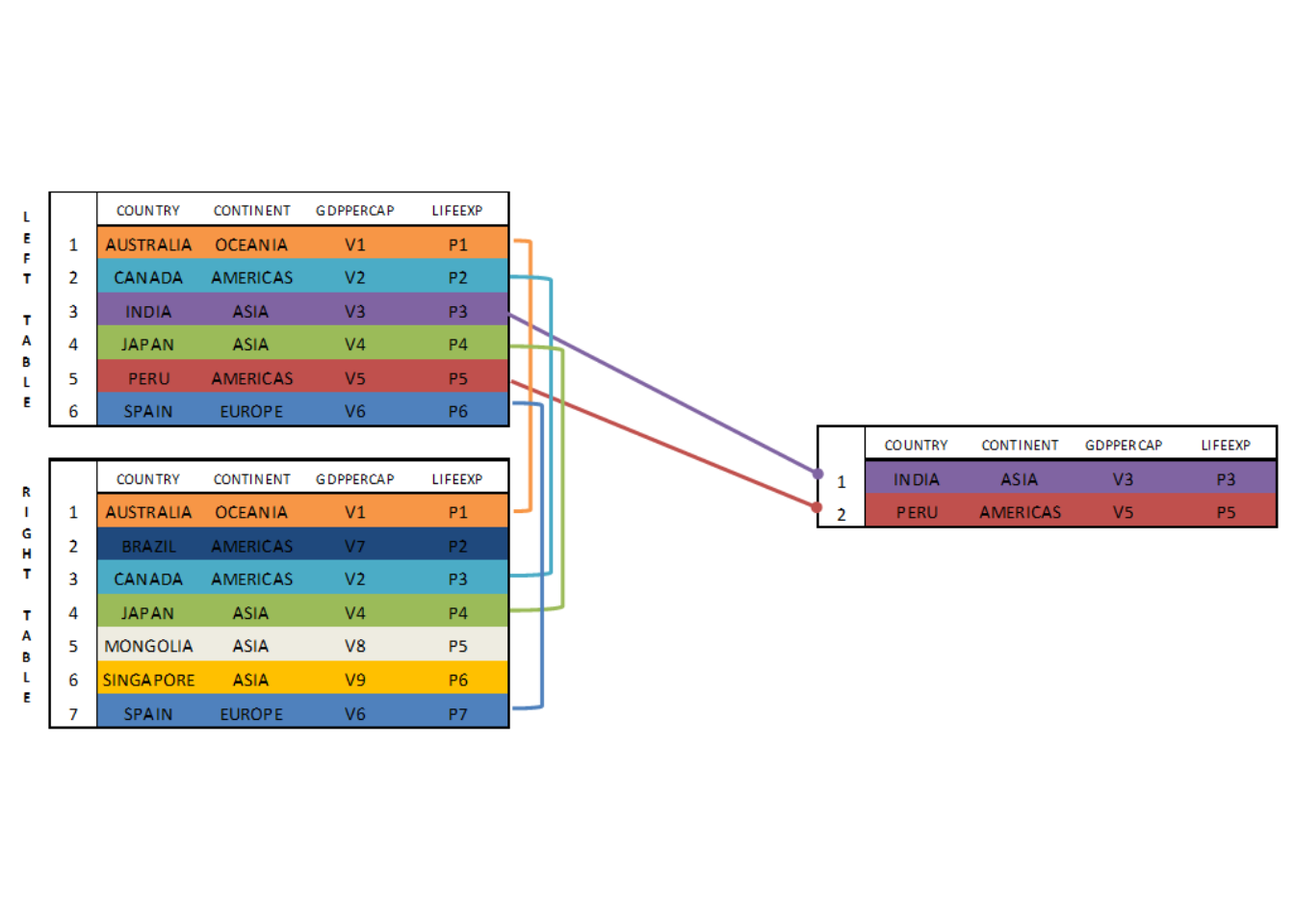
sqldf('SELECT country, continent, gdpPercap
FROM A
EXCEPT
SELECT country, continent, gdpPercap
FROM B')
## country continent gdpPercap
## 1 India Asia 2452.210
## 2 Peru Americas 7408.906
El comando análogo en R se denomina setdiff() y su operatividad se asemeja a las funciones union() e intercept() expuestas en los párrafos previos.
setdiff(A,B)
## # A tibble: 2 x 4
## country continent gdpPercap lifeExp
## <fct> <fct> <dbl> <dbl>
## 1 India Asia 2452. 64.7
## 2 Peru Americas 7409. 71.4
setdiff(B,A)
## # A tibble: 3 x 4
## country continent gdpPercap lifeExp
## <fct> <fct> <dbl> <dbl>
## 1 Brazil Americas 9066. 72.4
## 2 Mongolia Asia 3096. 66.8
## 3 Singapore Asia 47143. 80.0